最近看到了一个肿瘤免疫微环境的数据分析好文章,标题是:《Conserved pan-cancer microenvironment subtypes predict response to immunotherapy》,链接:https://doi.org/10.1016/j.ccell.2021.04.014,是德克萨斯大学MD安德森癌症中心的研究团队与2021年7月发表在:《Cancer Cell》杂志。
该数据挖掘的重点结论是:使用29个TME特征基因集合对肿瘤转录组队列可以无监督聚类确定了四种不同的微环境亚型:
• 免疫丰富、纤维化(IE/F);
• 免疫强化,非纤维化(IE);
• 纤维化(F);
• 免疫耗竭(D)
文章里面的knowledge-based functional gene expression signatures (Fges)就是TME特征基因集合,在附件有全部的基因信息:
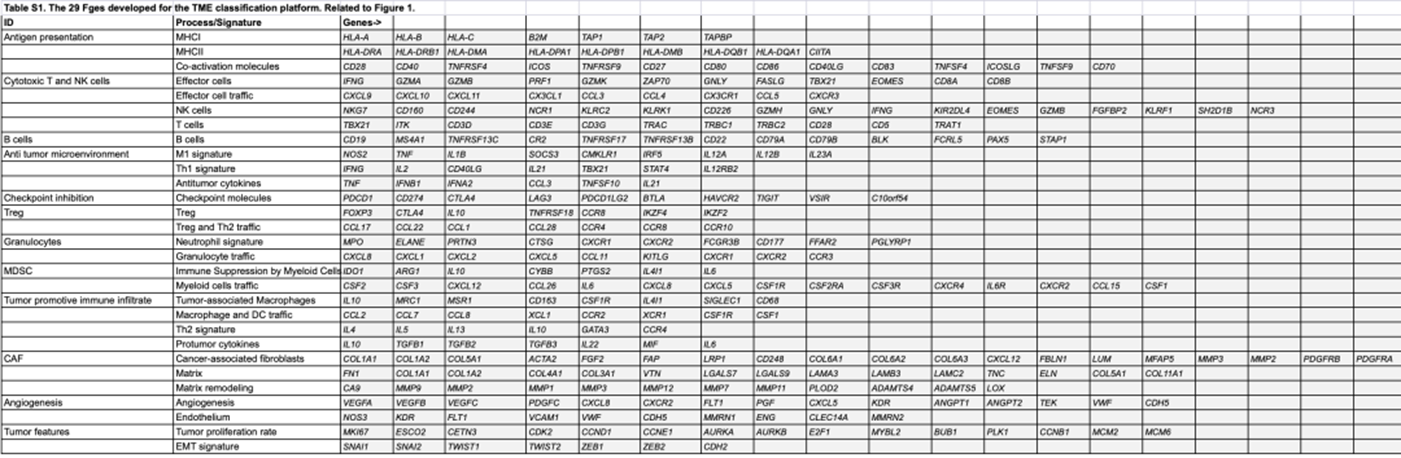
全部的基因信息
如果你想复用它,可以看文章的附件去复制粘贴哦:
• a manually curated list of 29 Fges was created covering known cellular and functional TME properties (STAR Methods, Table S1; Figures 1A and 1B).
• Each Fges was refined to include only genes associated with a specific cell type or biological process (Table S2, Figures 1C, S1A, and S1B).
这29个TME特征基因集合有一个简单的生物学意义分组:

生物学意义分组
首先进行泛癌打分
TME classification by transcriptomic analysis of >10,000 cancer patients identifies four distinct TME subtypes conserved across 20 different cancers.
如下所示,很容易下载TCGA的全部癌症的表达量矩阵,并且使用29个TME特征基因集合进行打分,打分的方法非常多其实,我们介绍过对单细胞表达矩阵做gsea分析的代码,以及单细胞转录组数据的批量GSVA代码大放送。
得分可以热图可视化如下所示:
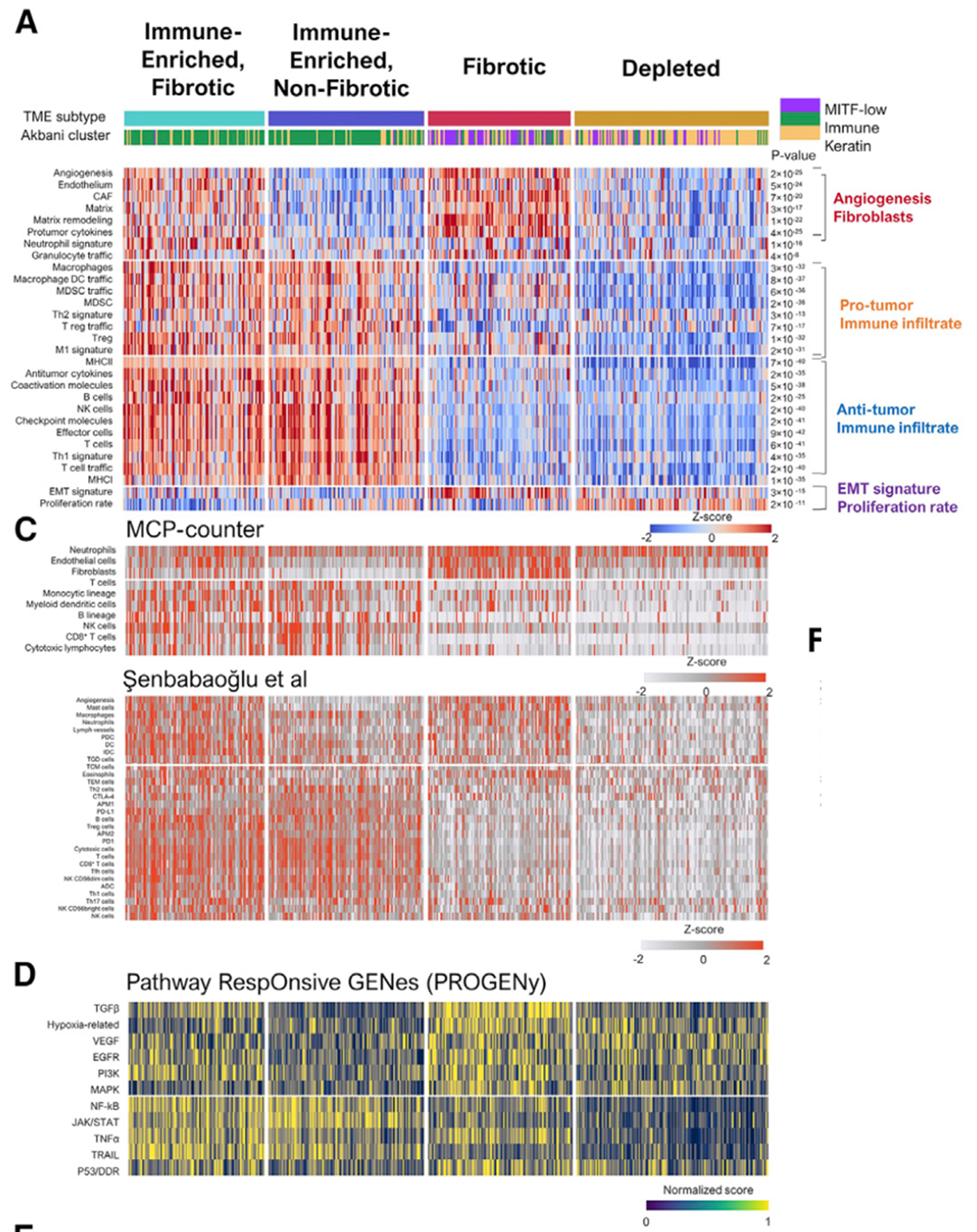
特征基因集合进行打分
比较好的区分成为了4类,是无监督的,另外一个展现方式是降维图,这里是tSNE的,如下所示:
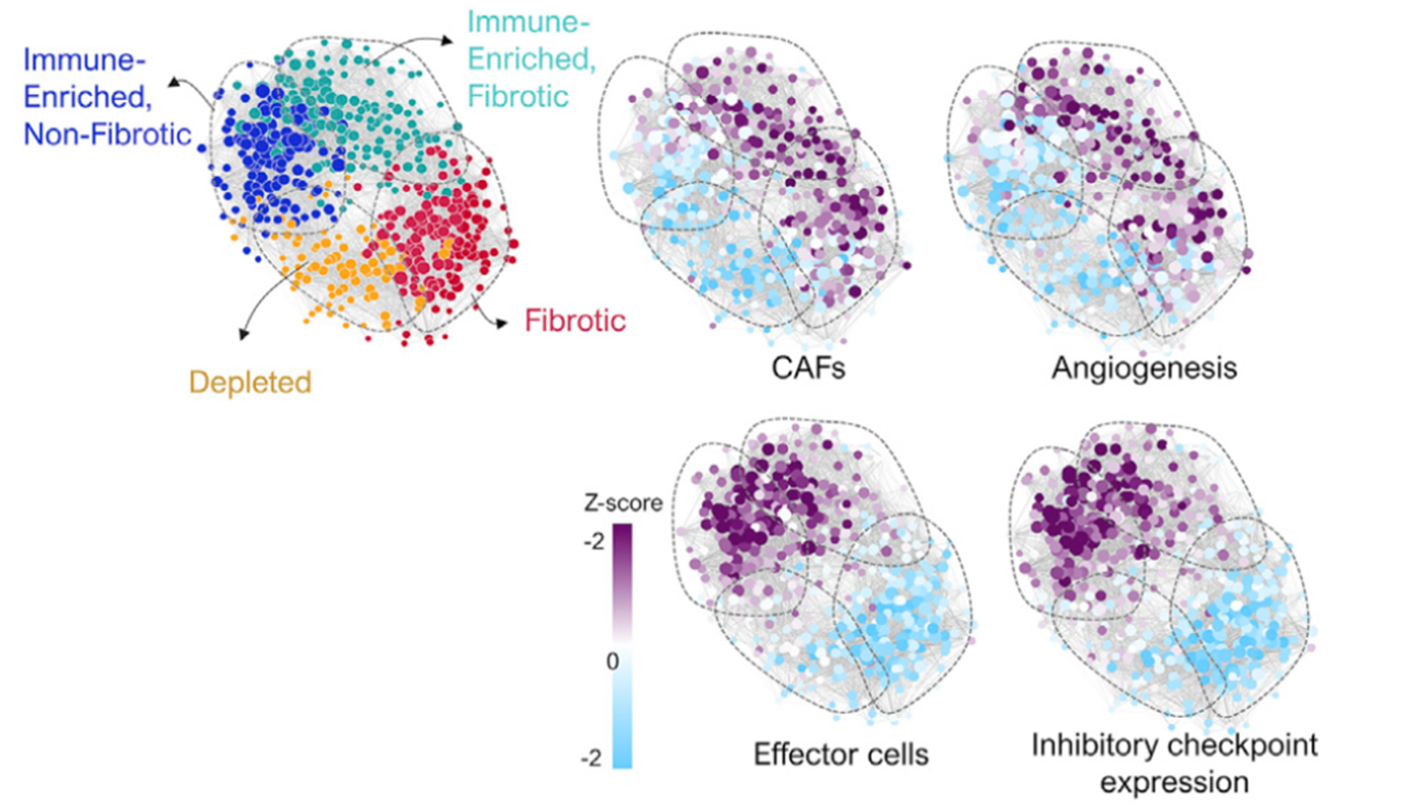
降维图
从热图和降维图,都可以很明显的看到1万多个肿瘤样品可以分成4组。对应前面的结论:使用29个TME特征基因集合对肿瘤转录组队列可以无监督聚类确定了四种不同的微环境亚型:
• 免疫丰富、纤维化(IE/F);
• 免疫强化,非纤维化(IE);
• 纤维化(F);
• 免疫耗竭(D)
后面就是丰富一下图表来描述这四种不同的微环境亚型,具有很好的跨癌症的应用场景,如下所示:
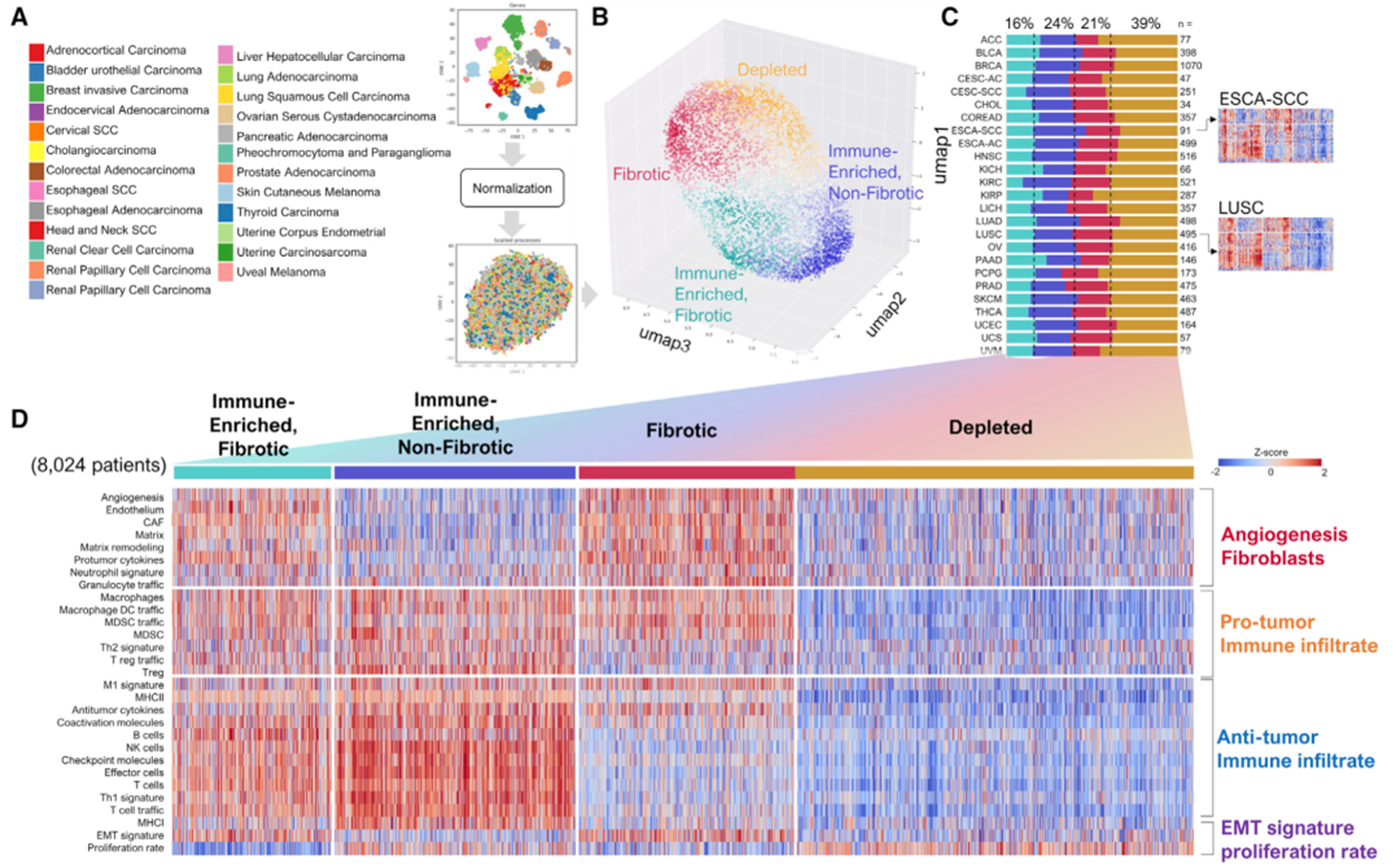
很好的跨癌症的应用场景
可以看到这样的4分类,在每个癌症都可以应用,而且后续还有举例的癌症内部4分类后的生存分析意义。
简单的生存分析当然不能满足《Cancer Cell》杂志要求,还可以进行免疫治疗队列的生存分析意义探索。
其它肿瘤免疫微环境背景知识
免疫系统在TME中具有多方面的作用,癌症免疫编辑的概念得到了许多研究证据的支持,癌症免疫编辑是免疫系统可以约束和促进肿瘤发展的过程,其通过称为消除(elimination),平衡(equilibrium)和逃逸(escape)的三个阶段进行。在这些阶段中,免疫系统改变肿瘤的免疫原性,并促进编辑的肿瘤逃避免疫识别。
其它类似的学说包括:
免疫炎症型,免疫豁免型和免疫沙漠型(2017)
2017年1月,Daniel Chen提出了一个新的癌症免疫表型,可以看作是免疫冷肿瘤与热肿瘤的升级版。Daniel Chen将肿瘤分为3种表型:免疫炎症型,免疫豁免型和免疫沙漠型。
免疫炎症型(immune-inflamed tumor)的肿瘤内部、基质、周围环境均有大量的免疫细胞浸润,处于激活或半激活状态。ICI容易在该免疫表型中发挥抗肿瘤效应。
免疫豁免型(immune-exclude tumor)肿瘤细胞周围也有大量免疫细胞存在,但免疫细胞不能渗透到肿瘤细胞内核,而是被限制在肿瘤细胞的外围基质。
免疫沙漠型(immnue-desert tumor)无论在肿瘤细胞内核和外围基质均缺乏T细胞。免疫沙漠型和免疫豁免型均被认为是非免疫感染表型。
见:Chen DS, Mellman I. Nature. 2017 Jan 18 541(7637) 321-330.
炎症和TMB的4象限分类法(2019)
2019年3月,Jake O’donnell的一篇综述将肿瘤微环境分为4个模式:
• 第1类TME肿瘤具有较高的肿瘤突变负荷(TMB)水平和炎症基因表达,提示可能存在持续功能性抑制的免疫应答。具有TMB水平或T细胞炎症基因特征的肿瘤似乎对ICI应答更好。
• 第2类TME肿瘤的TMB水平低,且缺乏炎症基因的表达。具有免疫无感或免疫细胞排斥表型的肿瘤可能属于2类TME。这类肿瘤患者如果接受免疫治疗,预后可能最差。
• 第3类TME肿瘤的TMB高于2类,但低于1类肿瘤的TMB,提示免疫原性新抗原可能是这些肿瘤的潜在靶点。但3类肿瘤中没有炎症基因表达提示3类肿瘤可能对免疫疗法不敏感。
• 第4类TME肿瘤中TMB水平低,但炎症基因表达高,这类肿瘤的PD-1/PD-L1免疫抑制机制不占主导地位,但是目前对免疫治疗的应答率尚不清楚。
见:O'Donnell JS, Teng MW, Smyth MJ. Nat Rev Clin Oncol. 2019 Mar 16(3) 151-167.
应该在单细胞水平看肿瘤免疫和基质
绝大部分的肿瘤研究单细胞研究我介绍过CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这个第一次分群规则是:
• immune (CD45+,PTPRC),
• epithelial/cancer (EpCAM+,EPCAM),
• stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
很明显,前面提到的2021年7月发表在:《Cancer Cell》杂志里面的29个knowledge-based functional gene expression signatures (Fges),可以在单细胞水平做一个泛癌研究。
转载自公众号《生信技能树》,如有侵权,请联系删除。