通常情况下,我们对基因的分组是统计学显著的上下调即可,就需要我们的实验设计是两个分组,比如癌症和正常,处理和对照等等。但是生物学实验设计往往是可以多元化,比如时间梯度或者浓度梯度的处理,都需要跟对照进行差异分析,这样的话,得到的基因集就非常多了。
而对基因的划分不同组别,还可以是根据表达量的相似性,代表性的方法有层次聚类、K-means聚类、WGCNA、Mfuzz等,其中Mfuzz是专门的做转录变化的时间趋势分析的方法,核心算法基于模糊c均值聚类(Fuzzy C-Means Clustering,FCM),关于它的用法我们很早以前就分享了笔记,见:使用Mfuzz包做时间序列分析。最近交流群有粉丝提问他看到了一个Mfuzz做转录变化的时间趋势分析后对每个趋势分组挑一个代表性基因,是发表在NaTure PLaNTS杂志的文章:《Jasmonate-mediated wound signalling promotes plant regeneration》,如下所示:
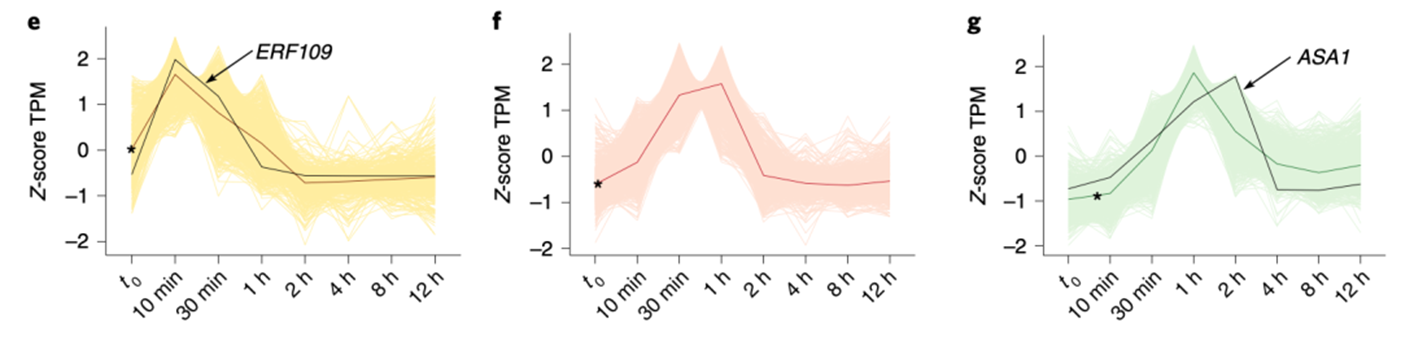
对每个趋势分组挑一个代表性基因
图例是e–g, RNA-seq data showing cluster-1 (e;n= 558 genes),-2 (f;n= 1,120 genes) and -3 (g;n= 982 genes) genes upregulated in response to wounding after the detachment of Col-0 leaves
其实就是unsupervised clustering by the fuzzy c-means algorithm。。。。implemented in the Mfuzz package ,简单看了看文章,好像是没有描述具体的唯一的显示在图上的基因是如何挑选到的,毕竟Mfuzz做转录变化的时间趋势分析后的每个趋势分组里面的都是成百上千个基因,如何挑选到最重要的的基因其实有很多方法。
这个文章的转录组数据在https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE120418,作者给出来了全部的24个样品的表达量矩阵文件,每个样品都是独立的文件:
GSM3400563_Coil_2_2h_S71_rsem_quals.genes.results.txt.gz 665.7 Kb
GSM3400564_Coil_2_2h_S72_rsem_quals.genes.results.txt.gz 664.9 Kb
GSM3400565_Coil_2_time0_S69_rsem_quals.genes.results.txt.gz 664.5 Kb
....
直接下载这个压缩包:https://ftp.ncbi.nlm.nih.gov/geo/series/GSE120nnn/GSE120418/suppl/GSE120418_RAW.tar
然后,我们批量读取其中的Col-0 leaves样品,如下所示:
GSM3400571 Col_0_10min_rep1
GSM3400572 Col_0_10min_rep2
GSM3400573 Col_0_12h_rep1
GSM3400574 Col_0_12h_rep2
GSM3400575 Col_0_1h_rep1
GSM3400576 Col_0_1h_rep2
GSM3400577 Col_0_2h_rep1
GSM3400578 Col_0_2h_rep2
GSM3400579 Col_0_30min_rep1
GSM3400580 Col_0_30min_rep2
GSM3400581 Col_0_4h_rep1
GSM3400582 Col_0_4h_rep2
GSM3400583 Col_0_8h_rep1
GSM3400584 Col_0_8h_rep2
GSM3400585 Col_0_time0_rep1
GSM3400586 Col_0_time0_rep2
可以看到,确实是0,10,30分钟,以及1,2,4,8,12小时的时间趋势分组。
下面我们简单的演示一下:
读取每个样品的表达量矩阵
d='GSE120418_RAW/'fs = list.files(d,pattern = '_Col_')fslibrary(data.table)tmp=fread(file.path(d,fs[1]))colnames(tmp)dim(tmp)#简单的学习了rsem_quals.genes.results.txt.gz的数据格式,自己去搜索RSEM软件哦lapply(fs, function(f){ print(dim(fread(file.path(d,f ))))})gene.count = do.call(cbind, lapply(fs, function(f){ ceiling(fread(file.path(d,f ),data.table = F)[,5]) }))head(gene.count)rownames(gene.count) = tmp$gene_idlibrary(stringr)colnames(gene.count) = str_split(fs,'_',simplify = T)[,4]
上面的代码需要你自己下载那个压缩包:https://ftp.ncbi.nlm.nih.gov/geo/series/GSE120nnn/GSE120418/suppl/GSE120418_RAW.tar,然后解压,就可以得到如下所示的表达量矩阵:
> gene.count[1:4,1:4]
10min 10min 12h 12h
AT1G01010 167 137 288 253
AT1G01020 386 422 346 328
AT1G01030 176 265 145 146
AT1G01040 1815 1245 1915 2466
可以看到这个是最原始的counts矩阵,而且每个时间点有3个样品,需要进行一些简单的处理,我这里的处理方式是:
dat <- log2(edgeR::cpm(gene.count)+1)dat[1:4,1:4]dim(dat)library(limma)avereps_df <- t(limma::avereps( t(dat) , ID = colnames(dat)))avereps_df[1:4,1:4]colnames(avereps_df)avereps_df = avereps_df[,c( "time0", "10min","30min" , "1h" , "2h", "4h" , "8h" , "12h" )]save(avereps_df,file = 'avereps_df.Rdata')load(file = 'avereps_df.Rdata')
这个时候的表达量矩阵就是被我简单的处理了,适合做下游分析啦,就是如下所示:
> avereps_df[1:4,1:4]
10min 12h 1h 2h
AT1G01010 2.019707 2.746182 3.437725 3.024456
AT1G01020 3.194309 3.022219 3.451913 3.481806
AT1G01030 2.429570 2.027529 4.783855 3.342235
AT1G01040 4.970931 5.553524 5.506124 5.682269
每个人处理原始的counts矩阵的方式不一样,希望你可以理解。
接下来就是运行Mfuzz包
希望你可以再次去阅读前面的笔记,见:使用Mfuzz包做时间序列分析,很简单的把前面的表达量矩阵拿去构建一个ExpressionSet的对象即可:
#首先安装R包并加载# BiocManager::install("Mfuzz")library(Mfuzz)library(limma)library(clusterProfiler)library(org.Hs.eg.db)library(ggplot2)library(ggstatsplot)library(tidyverse)## 2.1 Filtering----#去除表达量太低或者在不同时间点间变化太小的基因等步骤# Mfuzz聚类时要求是一个ExpressionSet类型的对象,所以需要先用表达量构建这样一个对象。eset <- new("ExpressionSet",exprs = avereps_df)#根据标准差去除样本间差异太小的基因eset <- filter.std(eset,min.std=0)# 10818 genes excluded.,不同的数据集去除的基因数量不一样eset ## 2.2 Standardisation----#聚类时需要用一个数值来表征不同基因间的距离,Mfuzz中采用的是欧式距离,#由于普通欧式距离的定义没有考虑不同维度间量纲的不同,所以需要先进行标准化eset <- standardise(eset) ## 2.3 Setting of parameters for FCM clustering----# Mfuzz中的聚类算法需要提供两个参数,#第一个参数为希望最终得到的聚类的个数,这个参数由我们直接指定#第二个参数称之为fuzzifier值,用小写字母m表示,可以通过函数评估一个最佳取值c <- 9m <- mestimate(eset) # 评估出最佳的m值cl <- mfuzz(eset, c = c, m = m) #聚类## 2.4 glimpse results----#在cl这个对象中就保存了聚类的完整结果,对于这个对象的常见操作如下cl$size #查看每个cluster中的基因个数cl$cluster[cl$cluster == 1] #提取某个cluster下的基因## cluster cores# membership values can also indicate the similarity of vectors to each other.esetcl.thres <- acore(eset,cl,min.acore=0.5) ## a posteriorihead(cl.thres[[1]])table(cl$cluster)unlist(lapply(cl.thres, nrow))
前面的表达量矩阵文件里面的基因是接近4万,但是我们的过滤仅仅是删掉了一万多表达量变化低基因,其实正常情况下我们应该是对基因进行筛选,比如先做差异或者提高过滤的阈值,比如剩下四五千个基因,比较适合去做:使用Mfuzz包做时间序列分析,或者WGCNA分析。
一个简单的可视化
前面的mfuzz分析我们是人为的指定了9个模块,而且我们给的基因数量实在是太多了,所以可视化有点丑:
library(RColorBrewer)color.2 <- colorRampPalette(rev(c("#ff0000", "Yellow", "OliveDrab1")))(1000)pdf('mfuzz_clusters_plot.pdf',height = 7,width = 12)mfuzz.plot(eset,cl,mfrow=c(3,3), new.window= FALSE, time.labels= colnames(eset) , colo = color.2)dev.off()
如下所示:
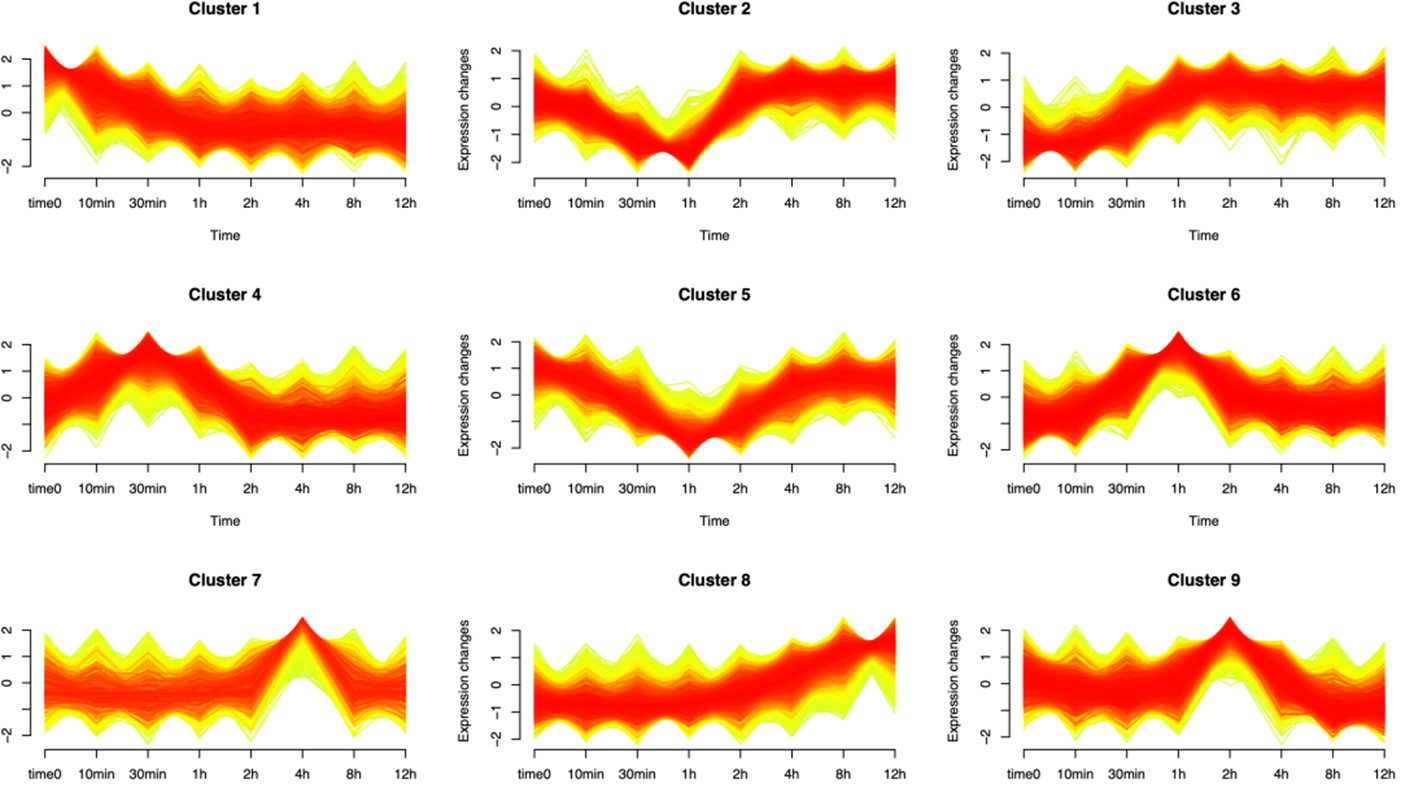
自己定义的9个模块
因为Mfuzz做转录变化的时间趋势分析后对每个趋势分组都是成百上千个基因,如下所示:
cl.thres <- acore(eset,cl,min.acore=0.5) unlist(lapply(cl.thres, nrow))# 1772 3113 1822 1931 2025 2659 786 2375 1640
其中acore函数帮助我们计算出来了每个趋势分组里面的成百上千个基因的重要性:
lapply(cl.thres, head)
[[9]]
NAME MEM.SHIP
AT1G01020 AT1G01020 0.6197867
AT1G01210 AT1G01210 0.7466814
AT1G01920 AT1G01920 0.8561513
AT1G01990 AT1G01990 0.9744499
AT1G02340 AT1G02340 0.5406831
AT1G02370 AT1G02370 0.8660542
我们只需要根据MEM.SHIP就可以挑选出来最具有代表性的基因啦,然后就可以把上面的9个模块,重新提取基因进行绘图。
转载自《生信技能树》,如有侵权,请联系删除。