本文由医信融合团队成员“陈浩然”撰写,已同步至微信公众号“医信融合创新沙龙”与“研究生学生信”,更多精彩内容欢迎关注!



Article name:SPEED2: inferring upstream pathway activity from differential gene expression
Journal: Nucleic Acids Res
Doi:10.1093/nar/gkaa236
IF:19.160
前言
这一切都要感谢他人的提问
在一个群里,有人发了一张这个图,其实图本身是很简单的热图,知道每个cluster的通路的正负相关之后,用ggplot2就能做出来。
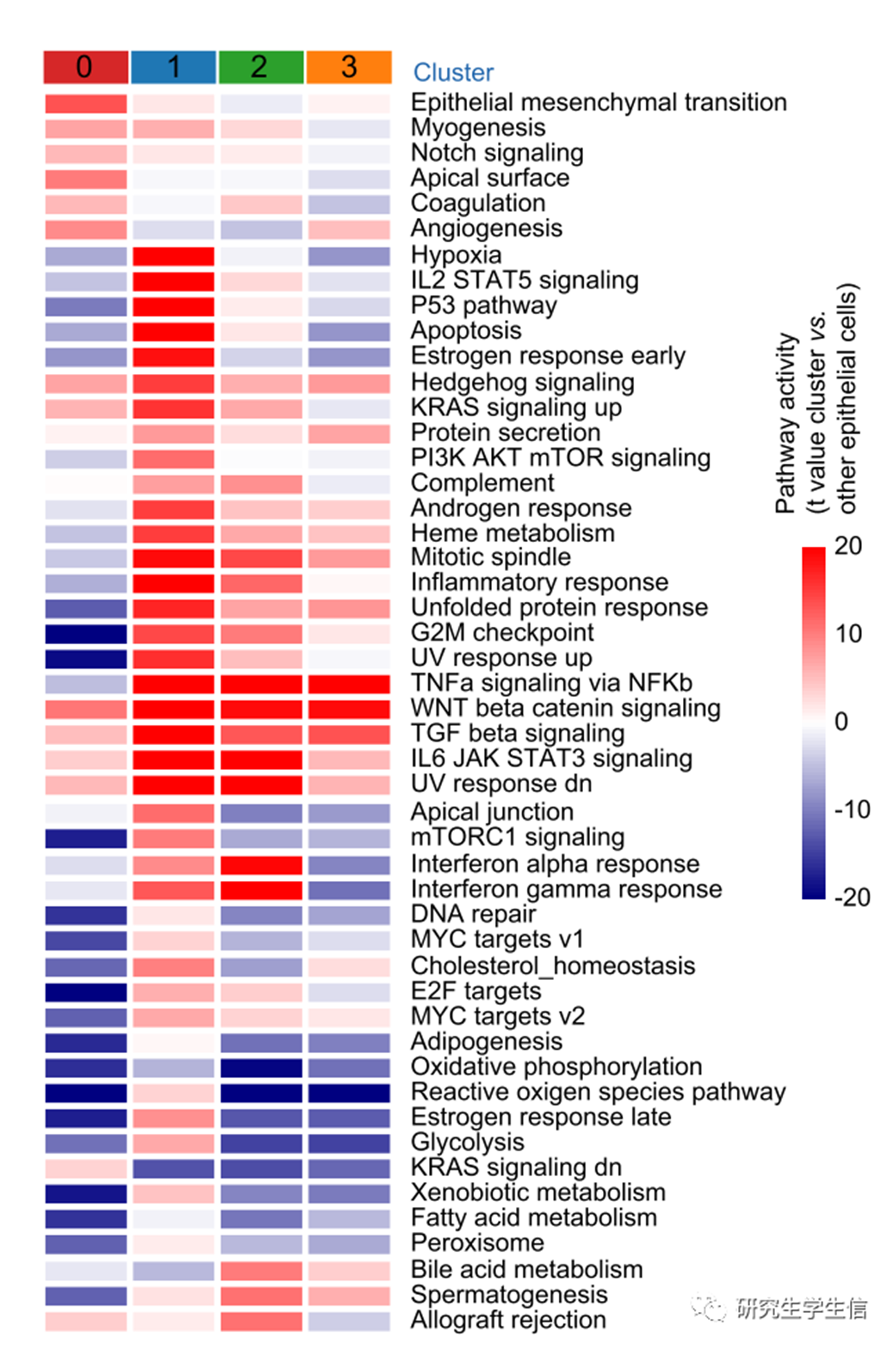
但是这个图例着实吸引了我,pathway activate,众所周知一个通路的激活与多种因素有关,而不仅仅是上下调关系,连忙问文章出处。可惜最终了解到,这仅仅也是基因集富集分析的一种,pathway activate也只是博人眼球的,它仅仅是推断了差异基因与通路的正负相关,而非真正的激活。
抱着好奇检索了一下是否有相关文献真正做pathway activate研究的,还真有!
也就是文章开头那篇文章。
文章细节就省略了,一句话解释就是基于**扰动数据训练的模型,对基因在通路中的富集情况进行评分、判断激活or抑制。**
而操作起来也很简单
输入文件为diff.txt
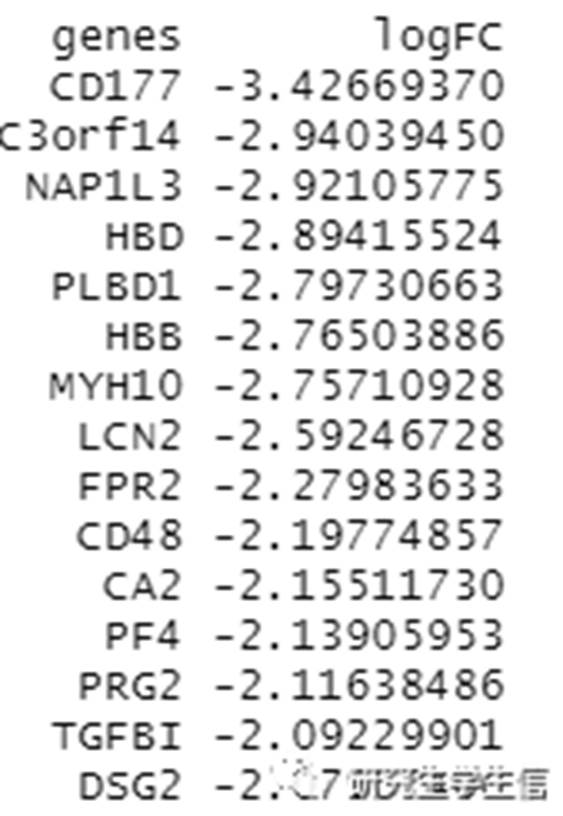
diff.txt
放在你的工作目录中,运行下面脚本就行了
setwd("")#设置你的工作目录
library(devtools)
install_github("molsysbio/speed2")
library(speed2)
diff <- read.table("diff.txt",sep = "\t",header = T)
testset=diff[,1]
ret = speed2run(testset,shuffle = F)
gg = speed2plot(ret$df_stattest, ret$df_rankcoords, test="chi2")
gg
然后就可以收获一张不太美丽但很实用的图片
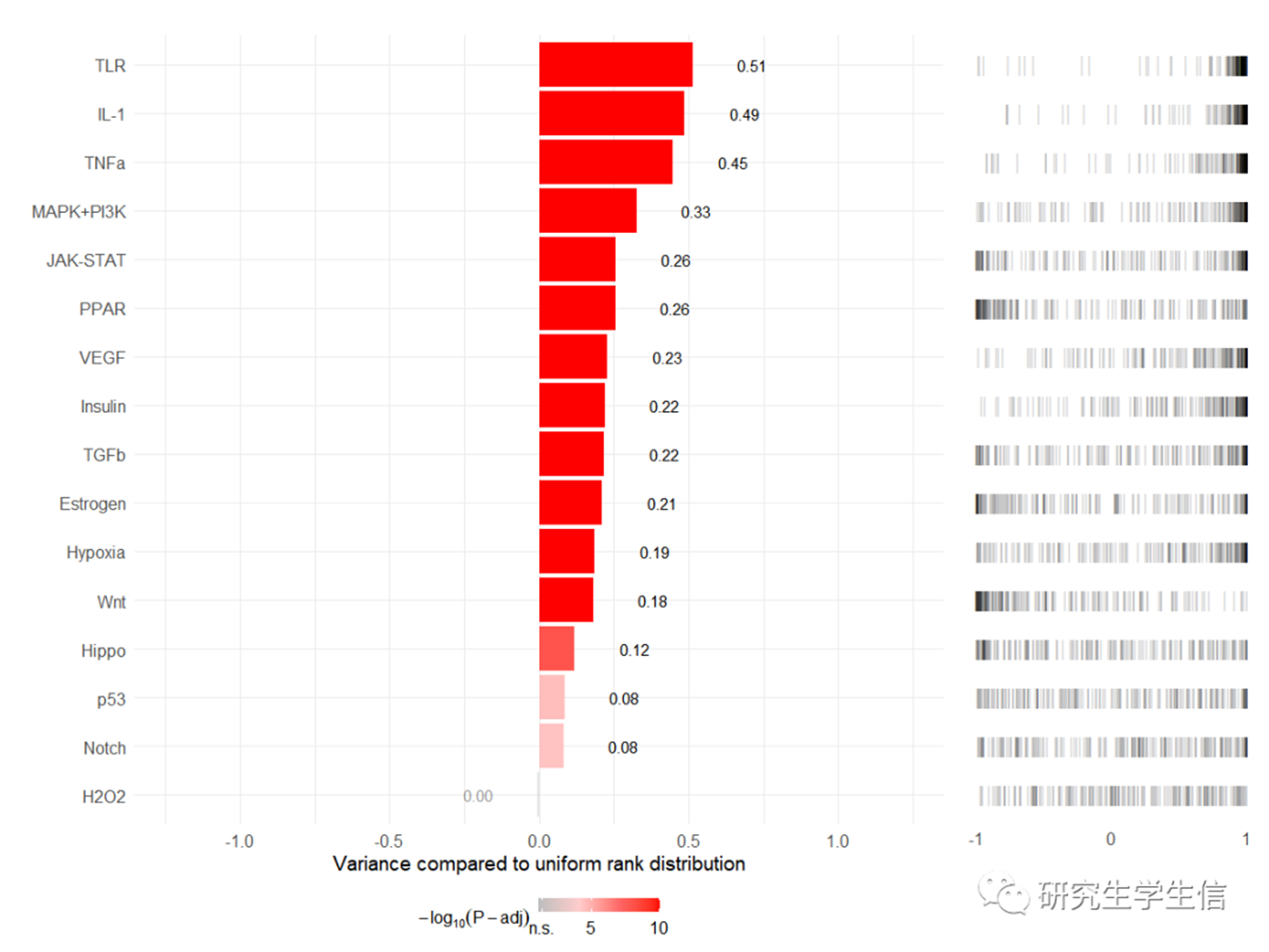
可以看到我的数据集里有一些通路是激活的,也有个问题昂,就是speed2只能做已有的16个数据集(也不是、只是这16个最准确)。按需使用即可。
还有一个问题就是
这图确实太丑了
但是我们可以自己用原始数据来画一个
> ret
$df_stattest
# A tibble: 16 × 6
speed2 p_ztest ztest_reg p_chi2test mean_rank chi2TestStatistic
<chr> <dbl> <chr> <dbl> <dbl> <dbl>
1 TLR 3.58e-143 UP 0 0.850 0.846
2 IL-1 4.11e-129 UP 0 0.838 0.820
3 TNFa 4.90e-103 UP 0 0.764 0.780
4 Insulin 6.20e- 42 UP 0 0.531 0.553
5 VEGF 1.18e- 41 UP 0 0.533 0.562
6 MAPK+PI3K 1.46e- 41 UP 0 0.530 0.659
7 Wnt 1.32e- 30 DOWN 0 -0.456 0.513
8 Hypoxia 1.29e- 13 UP 0 0.292 0.519
9 TGFb 3.52e- 8 UP 0 0.217 0.551
10 PPAR 1.23e- 6 DOWN 0 -0.190 0.589
11 JAK-STAT 1.90e- 6 UP 0 0.186 0.590
12 H2O2 6.34e- 4 UP 1 0.133 0.329
13 Notch 3.89e- 3 UP 0.0000481 0.108 0.415
14 Estrogen 2.87e- 2 DOWN 0 -0.0769 0.543
15 Hippo 3.53e- 2 DOWN 0.0000000113 -0.0740 0.451
16 p53 1.84e- 1 UP 0.0000310 0.0366 0.418
$df_rankcoords
# A tibble: 3,192 × 2
speed2 rank
<chr> <dbl>
1 Estrogen -0.903
2 Estrogen -0.997
3 Estrogen 0.866
4 Estrogen -0.426
5 Estrogen -0.559
6 Estrogen -0.450
7 Estrogen 0.0554
8 Estrogen 0.341
9 Estrogen 0.624
10 Estrogen -0.712
# … with 3,182 more rows
# ℹ Use `print(n = ...)` to see more rows
$unknown_items_geneset
numeric(0)
$unknown_items_backgrund
character(0)
可以看到,ret中储存了原始的绘图所需信息,上面的图其实就是geom_bar+geom_line。俩函数再加一点theme()
如果想知道这里边每个字段代表什么意思,可以运行以下代码进行查看
vignette("speed2")
敬请批评指正!

图文:陈浩然
本文编辑:莫状