本文由医信融合团队成员“张力中”撰写,已同步至微信公众号“医信融合创新沙龙”与“表观精准治疗”,更多精彩内容欢迎关注!


生信分析中,gene symbol 与 geneID 的互相转换是使初学者十分头疼的问题,今天给大家介绍的方法,不用专门下载人类基因组注释文件,不用使用复杂的代码,就可以实现 gene symbol 与 geneID 之间的灵活转换,一起试试吧!
前期准备
安装 biomaRt 包
#在R语言中运行
if (!require("BiocManager",quietly = TRUE))
install.packages("BiocManager")
BiocManager::install("biomaRt")
#查看安装情况
library(biomaRt) #加载包
listMarts() #查看目标数据库(mart),主要是版本号version,会定期更
选择数据库和版本号
ensembl <- useEnsembl(biomart ="ensembl",dataset = "hsapiens_gene_ensembl",mirror ="useast")
#参数dataset可更换,“人”选择"hsapiens_gene_ensembl",“小鼠”选择”mmusculus_gene_ensembl”
#官网链接http://ensemblgenomes.org/
#listDatasets(ensembl)可查找基因组名称
一步完成转换
setwd("C:\\Users\\Desktop\\2_symbol转换ID")
expMatrix <-read.table(file="ensembl.txt", header = T ,sep ="\t",row.names = NULL)
test_genes<-expMatrix[,c(1)]
test_genes
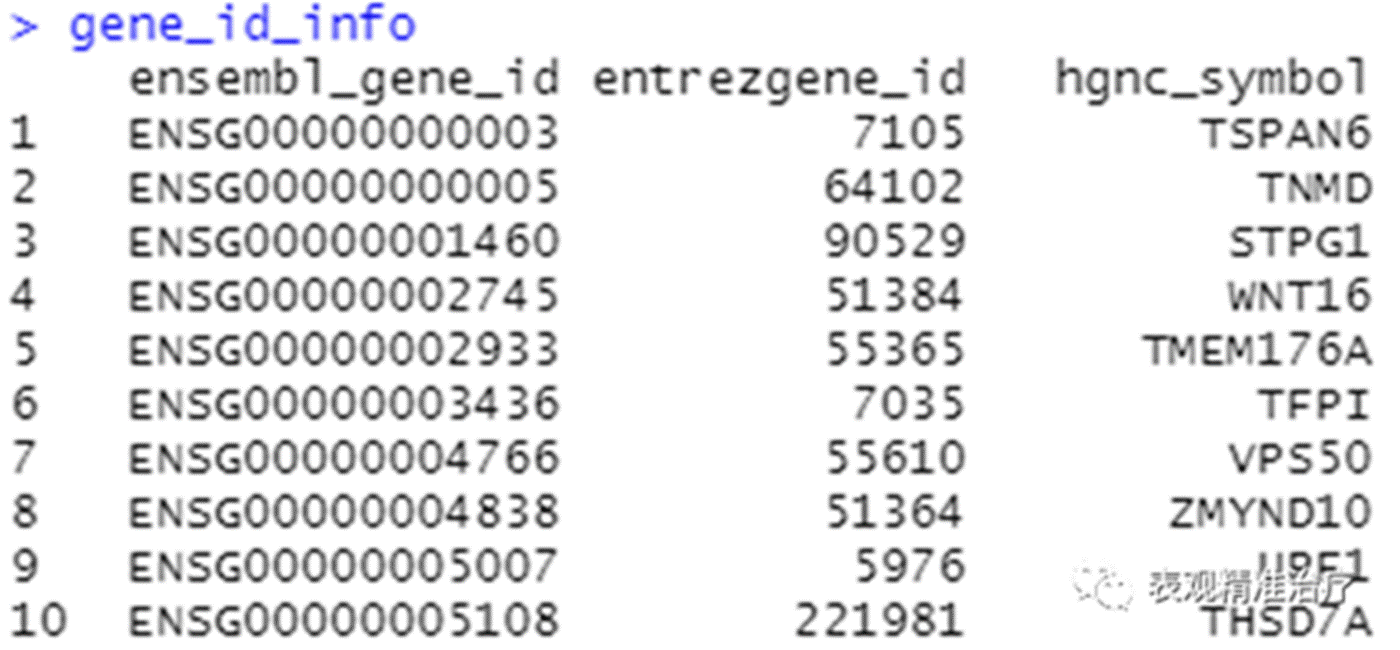
gene_id_info<- getBM(attributes=c("ensembl_gene_id","entrezgene_id","hgnc_symbol"),
filters ="ensembl_gene_id",
values=test_genes , mart=ensembl)
##参数说明:
#attributes参数是你需要返回的gene信息,需要什么类型都写上(listAttributes()可以查看所有gene attribute类型)
#filters参数是你当前能提供的gene的ID类型,而values的值就是ID类型的具体值,一般是一个向量
#ensembl_gene_id:Ensembl ID,存储数据最常用的ID号
#entrezgene_id: ncbi ID
#hgnc_symbol: gene symbol,也就是基因名
还可以标注基因在哪一条染色体上(如无必要,可不运行)
gene_id_info<-getBM(attributes=c("ensembl_gene_id","entrezgene_id","hgnc_symbol"),filters ="chromosome_name",values=c(1:22,'X','Y'),mart= my_mart)
annotation<-merge(expMatrix,gene_id_info, by.x = 'ensembl_gene_id', by.y = 'ensembl_gene_id' ,all.x = TRUE,sort = TRUE) #合并数据及ID
annotation<-annotation[complete.cases(annotation),] #去除空值

保存结果
write.table(gene_id_info,file ="annotation.txt",sep = "\t",row.names = F,col.names =T,quote = F)
图文:张力中
本文编辑:李晨龙