因此,这个数据库还是灰常良心和稳定的,自2014年发布以后,一直有人在投入维护,并且在行业内形成一个良性循环,诸如提供circRNA的命名规则,提供UCSC gemone browser信息等。
功能介绍
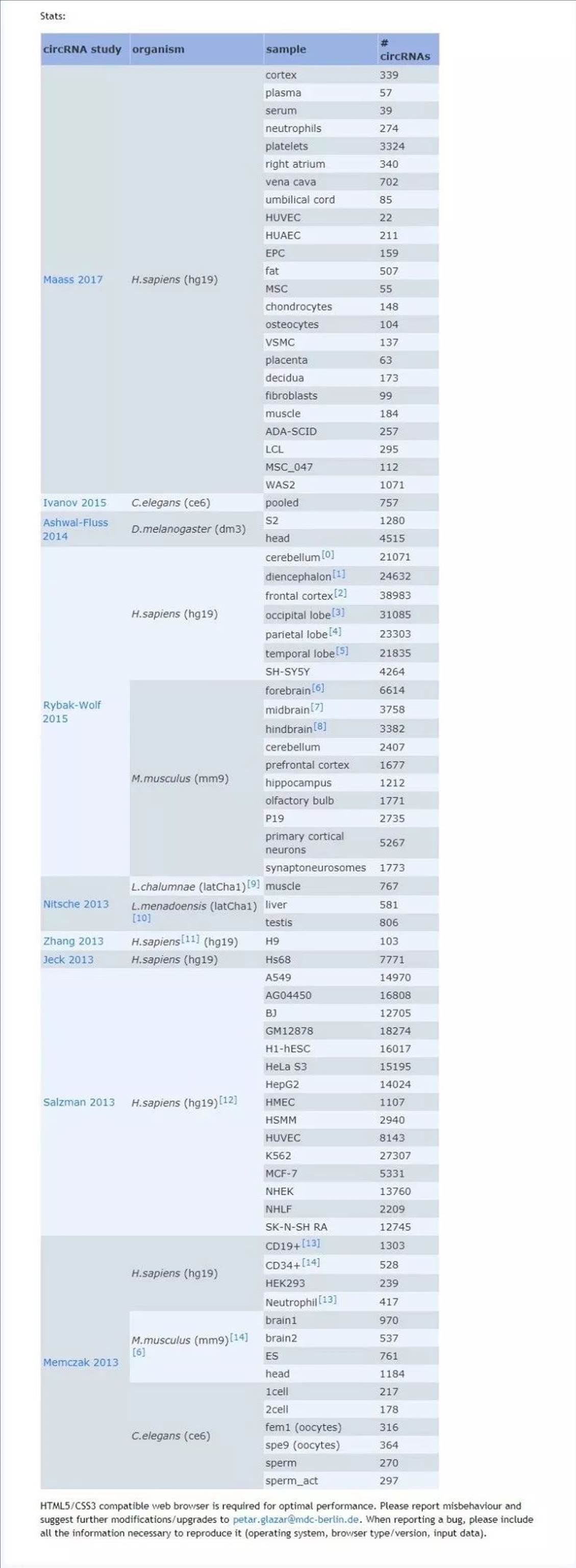
首先我们来看数据库上涵盖的数据信息,截至发稿,主要包括的测序物种有Homo sapiens, Mus musculus, Caenorhabditis elegans, Latimeria等。
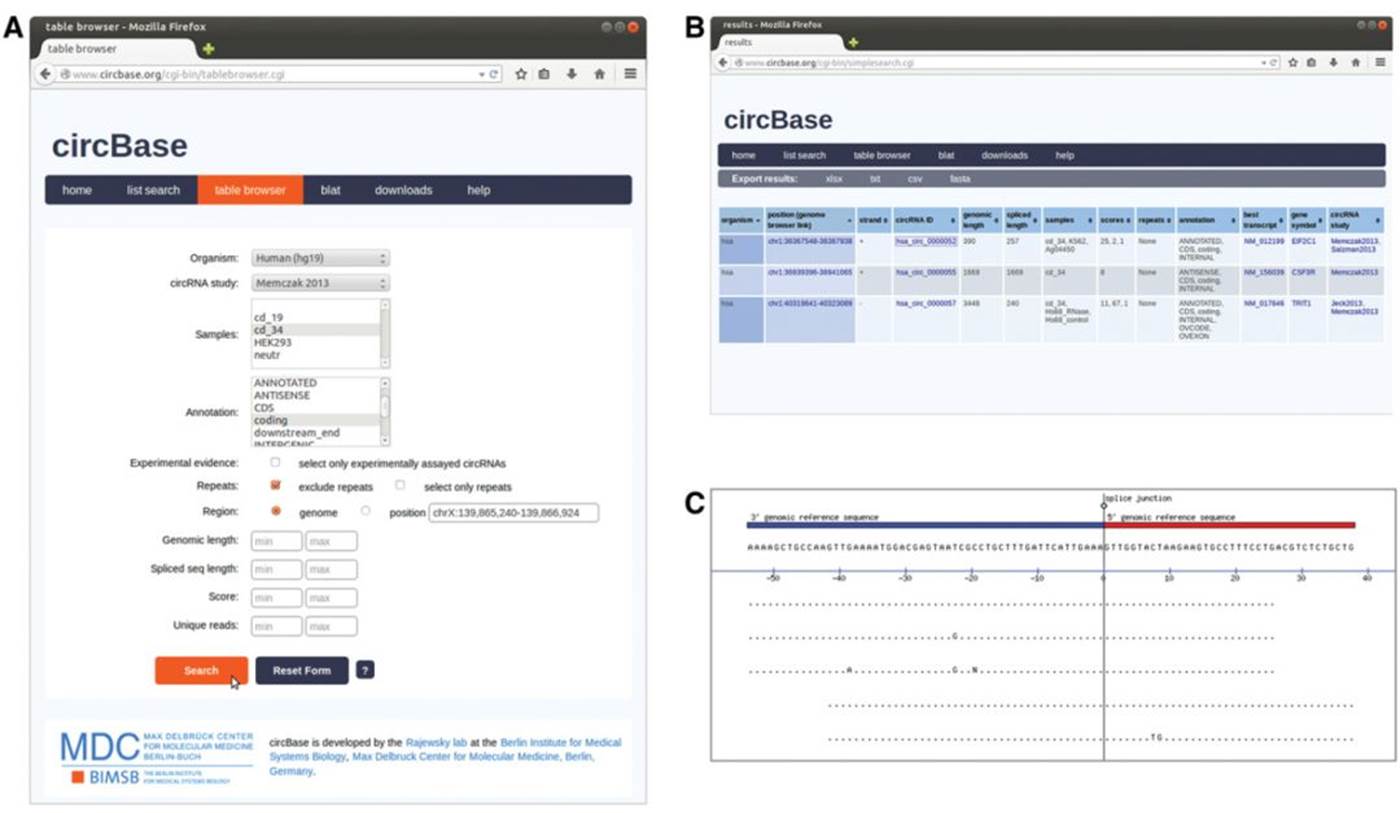
circBase获取数据主要有以下三种方式:
• simple search
• List search
• Table browser
涵盖绝大多数简易搜索的词条,包括输入DNA or RNA序列后,使用Blat获取反向互补序列或者获得包含circular junctions的circRNA序列;符合规则的输入基因组位置能够获得circRNA overlapping repeats等一系列信息;当然也可以通过简单的关键词搜索,获得标准格式的12-column BED文件。简单的搜索内容如下图:
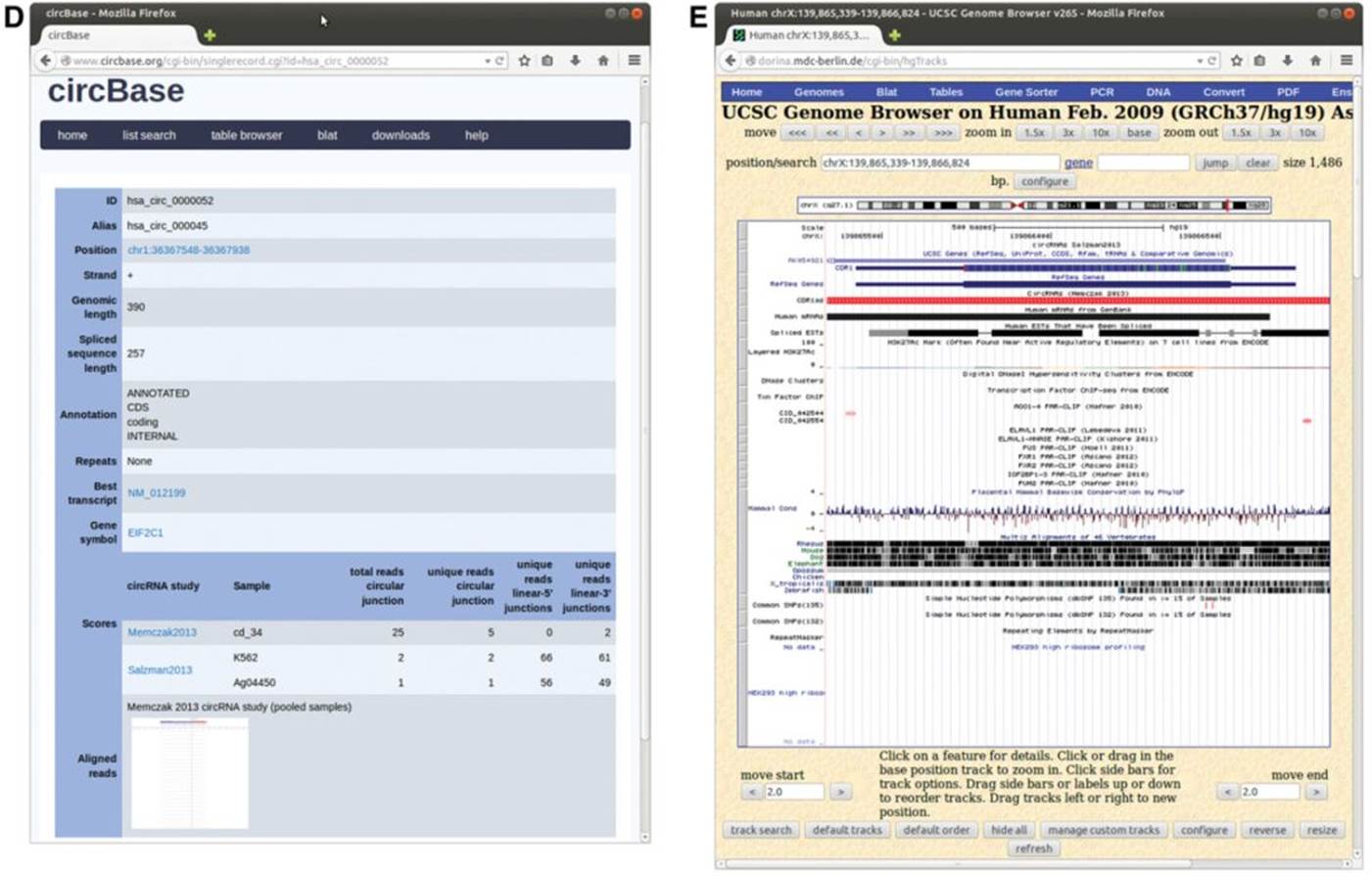
下载数据格式包括.xlsx, tab-separated .txt以及FASTA format,FASTA文件通常以tar.gz打包。在数据库上,作者非常友好的将如何发现circRNA所需要的所有代码(其实就是find_circ),以及用于测试的自测RNA-seq数据放入downloads页面,详细内容可见README。
注意事项
关于数据标签的展现形式,比如circBase上hg19 for H. sapiens, mm9 for M. musculus and ce6 for C。因此可以通过liftOver上进行转换。
本文转载自《生信草堂》。如有侵权,请联系删除。