数据库(Database,简称DB)是长期储存在计算机内、有组织的、可共享的数据集合。数据库中的数据按一定的数据模型组织、描述和储存、可为各种用户共享,此外数据库还具有冗余度较小、数据独立性较高、易扩展等特征。数据库的建设规模、数据库信息量的大小和使用频度已成为衡量一个国家信息化程度的重要标志。本期给大家分享笔者学习的The CancerImmunome Atlas**(TCIA数据库)**,这个数据库主要是根据TCGA的二代测序数据开发出来的。
TCIA数据库的全称是The Cancer Immunome Atlas(癌症免疫图谱)
一般来说我们利用Bing搜索“TCIA”都能够找到
大家记得和昨天的The CancerImaging Archive**(TCIA)数据库**的学习笔记区分开哦
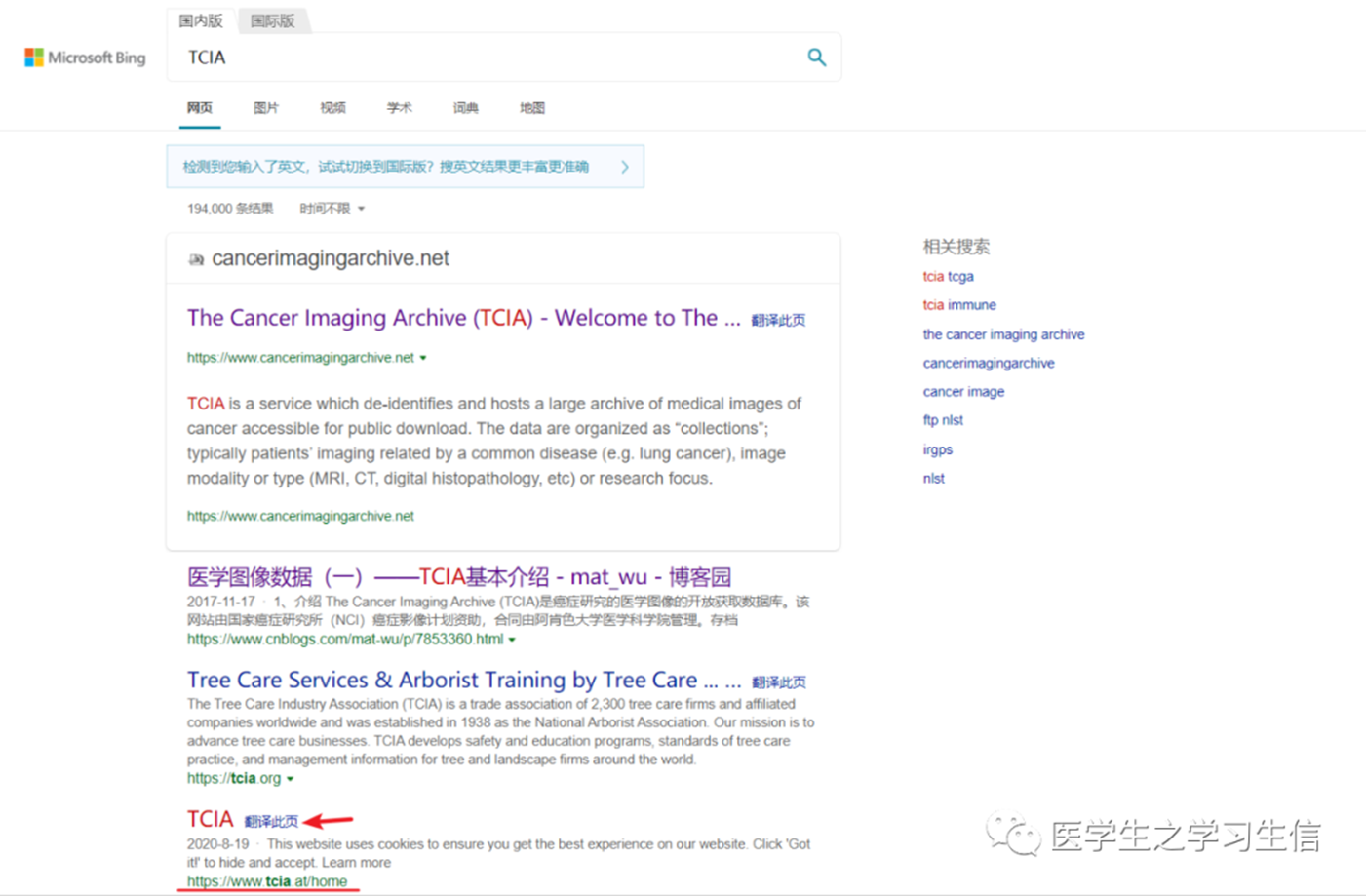
需要注意的是,bing搜索结果的第一条并不是我们要的TCIA数据库,下面才是我们要找的TCIA数据库
网站网址:https://tcia.at/home
 网址的首页是这样↑
网址的首页是这样↑
TCIA基于TCGA数据开发,不同的是TCIA只提供了20个癌种的免疫数据分析
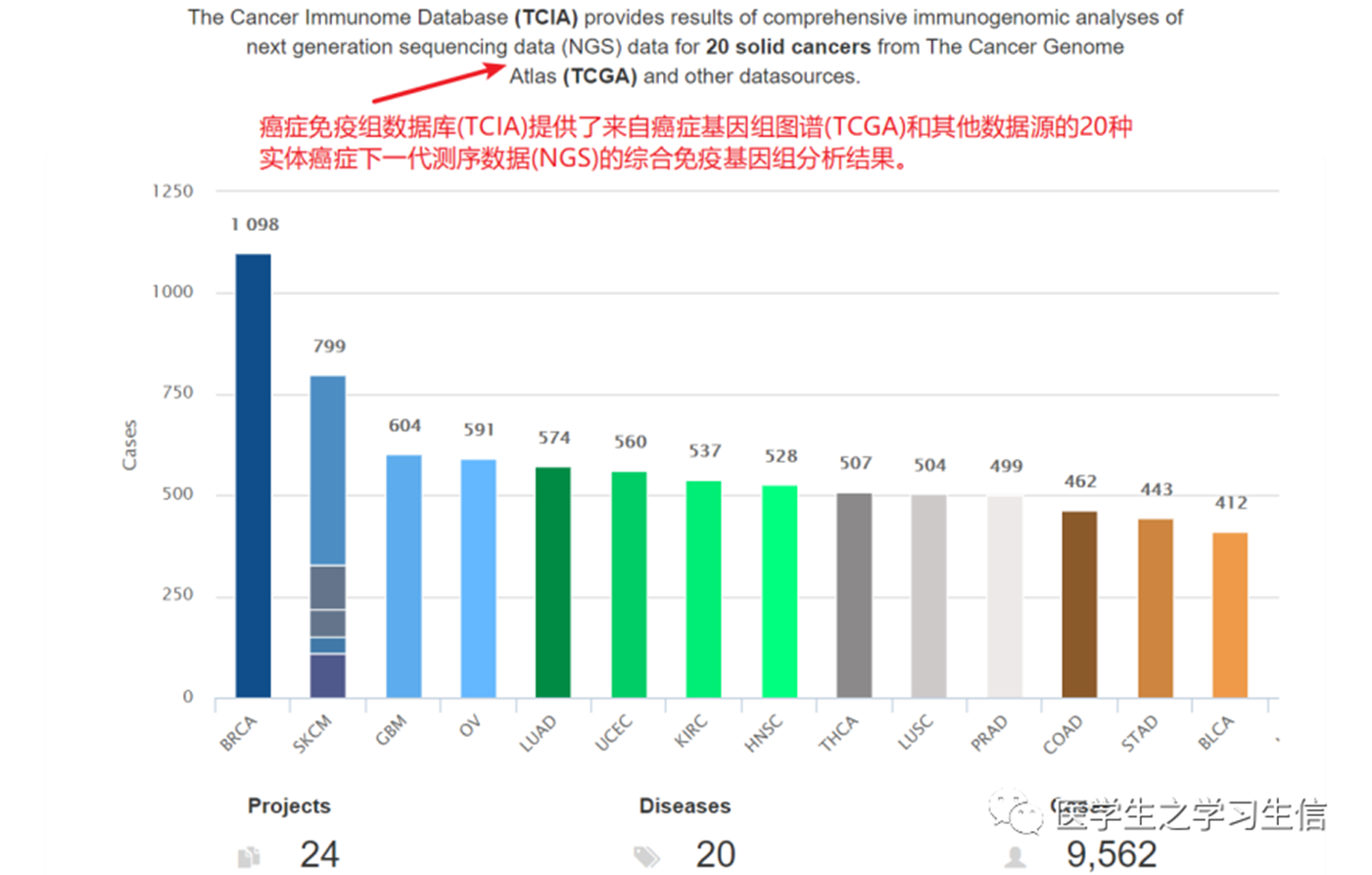
这里共20个癌种,点击每个柱子进去就可以分析对应的癌种↑
(移动光标还可以看到数据来源,以SKCM为例,可以看到它的数据来源不止TCGA,还有Rizvi 2017等)
随机选择一种癌症
 我们首先可以观察到界面上方有很多模块↑
我们首先可以观察到界面上方有很多模块↑
01 Patient list
TCIA分别对每个病人进行分析,数据也提供下载,我们可以看到提供了ID、疾病、性别和年龄信息。
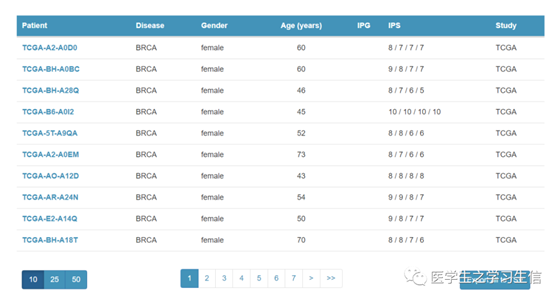 可以看到很多不同的病例↑
可以看到很多不同的病例↑
 点击病例,会展开其详细信息
点击病例,会展开其详细信息
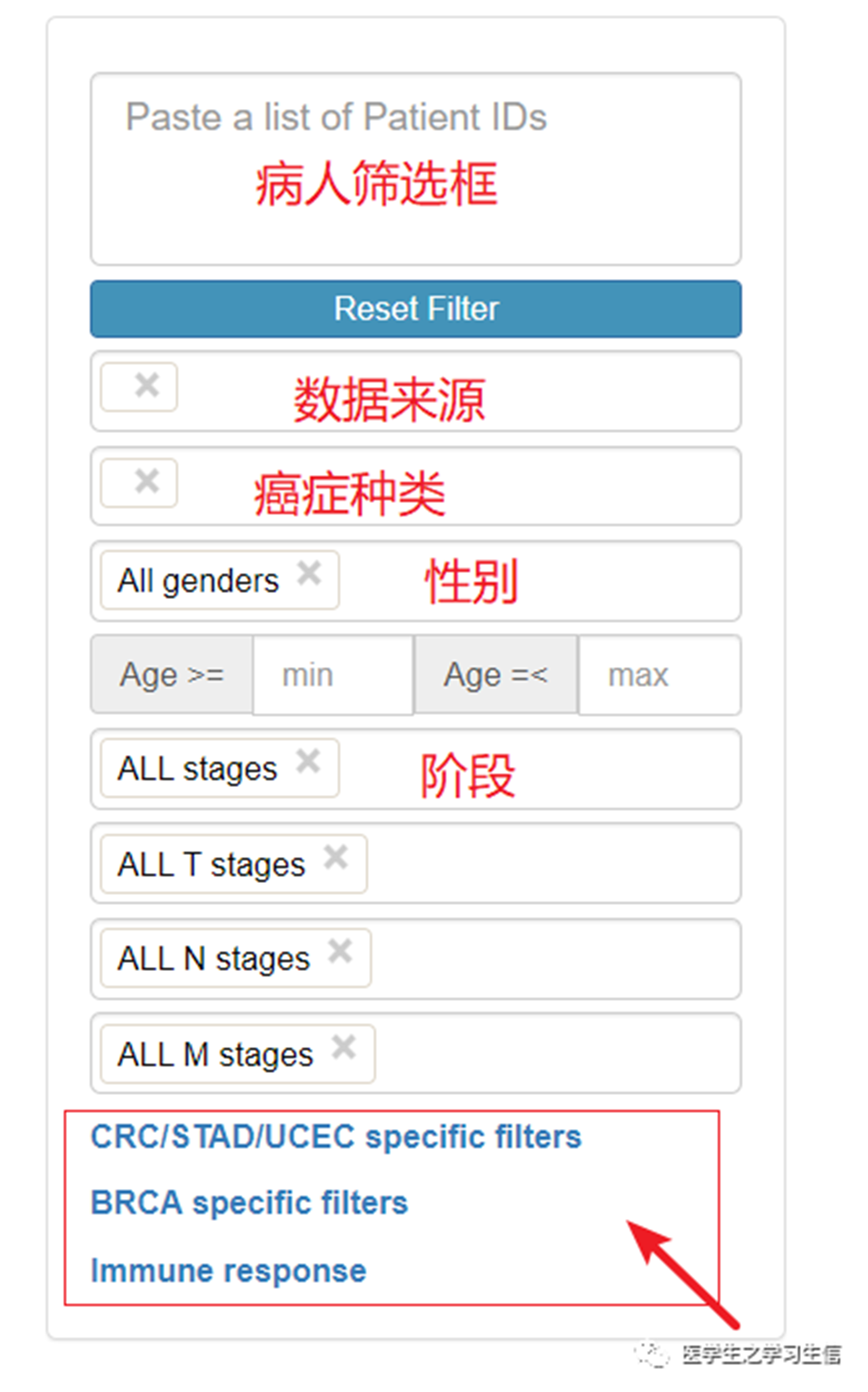
页面的左侧我们可以看到有一个病例信息筛选框↑
红色框内是一些特定种类癌症的特征筛选,如CRC/STAD/UCEC specific filters有MSI、甲基化等。
02 Gene Expression
在左侧的选择栏里筛选病人和基因后,可对单个基因进行差异分析和生存分析,值得注意的是这些基因都是和免疫相关的基因。
 注意,进入Gene Expression模块以后,网页会提示你先选择相应的基因,也就是红色箭头所指的框会自己变红↑
注意,进入Gene Expression模块以后,网页会提示你先选择相应的基因,也就是红色箭头所指的框会自己变红↑
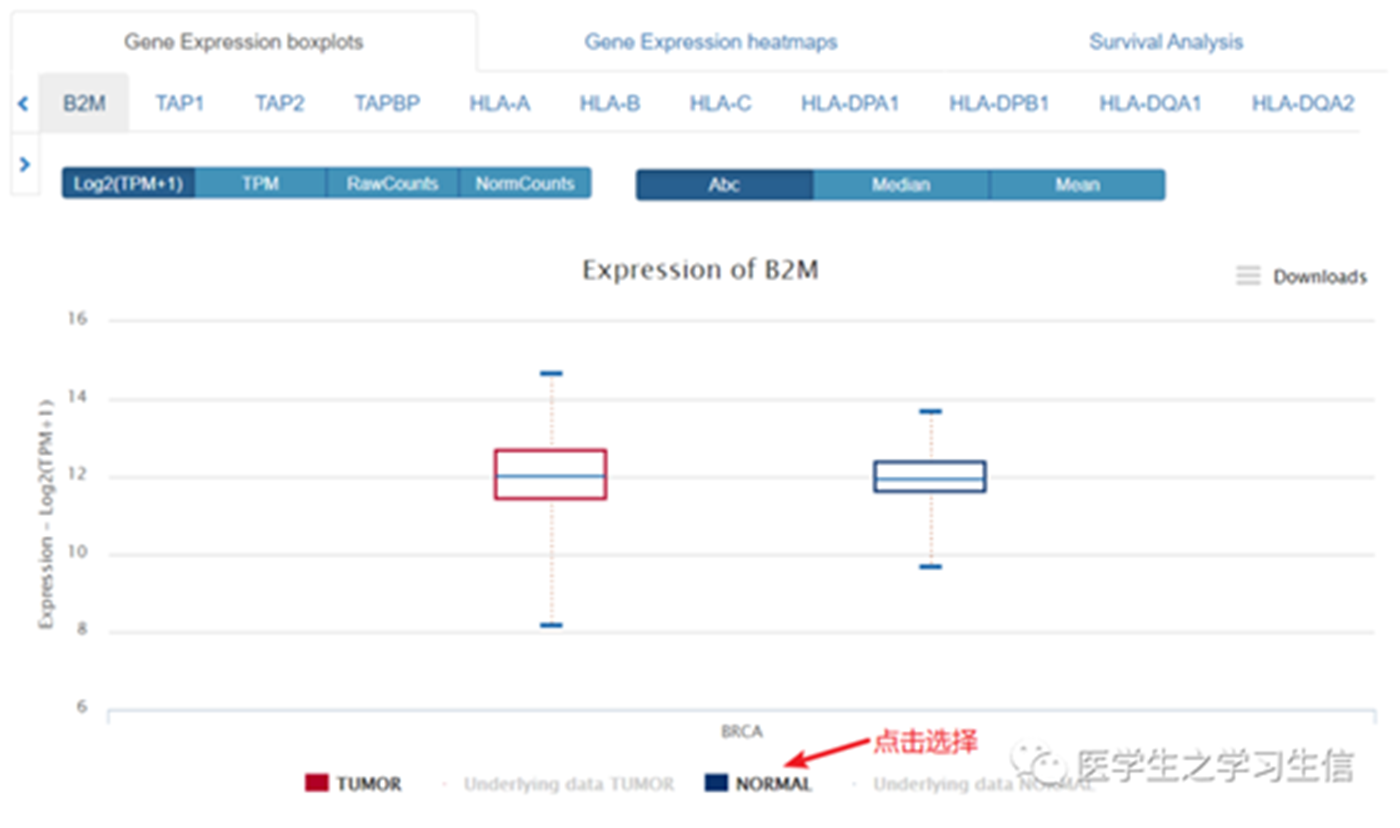 ↑Gene Expression boxplots
↑Gene Expression boxplots
 ↑Gene Expression heatmaps
↑Gene Expression heatmaps
 ↑Survival Analysis
↑Survival Analysis
03 Cell Fraction
这个模块提供不同免疫细胞比例的展示。
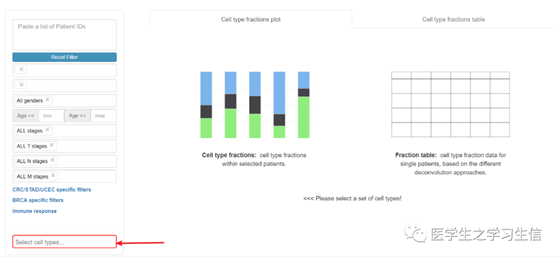 与Gene Expression模板类似,网页也会提醒你先选择相应的基因↑
与Gene Expression模板类似,网页也会提醒你先选择相应的基因↑
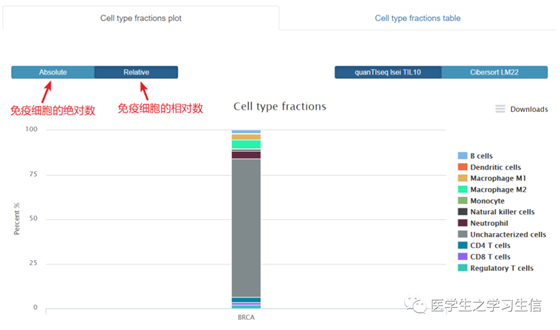 Absolute和Relative表示免疫细胞的绝对数和相对数,是CIBERSORT的不同计算方法结果↑
Absolute和Relative表示免疫细胞的绝对数和相对数,是CIBERSORT的不同计算方法结果↑
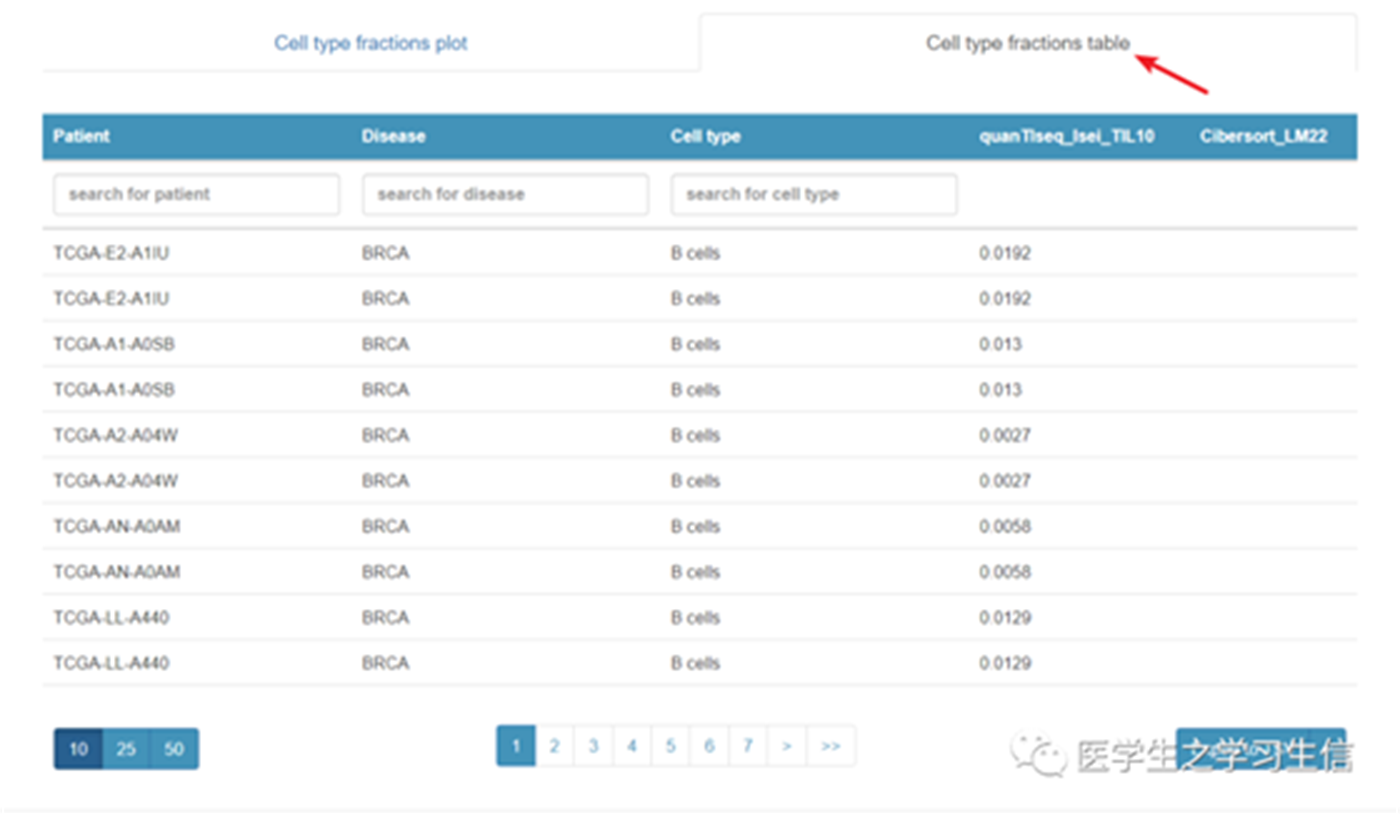 table栏提供快捷的搜索↑
table栏提供快捷的搜索↑
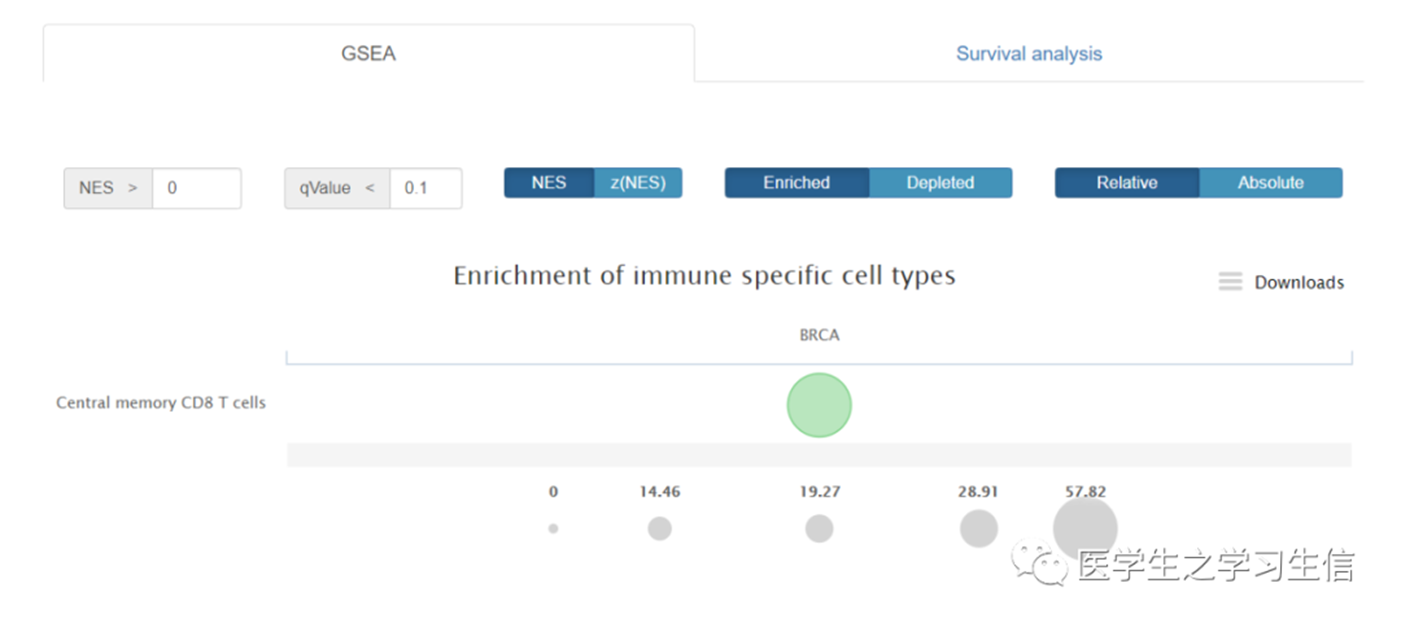
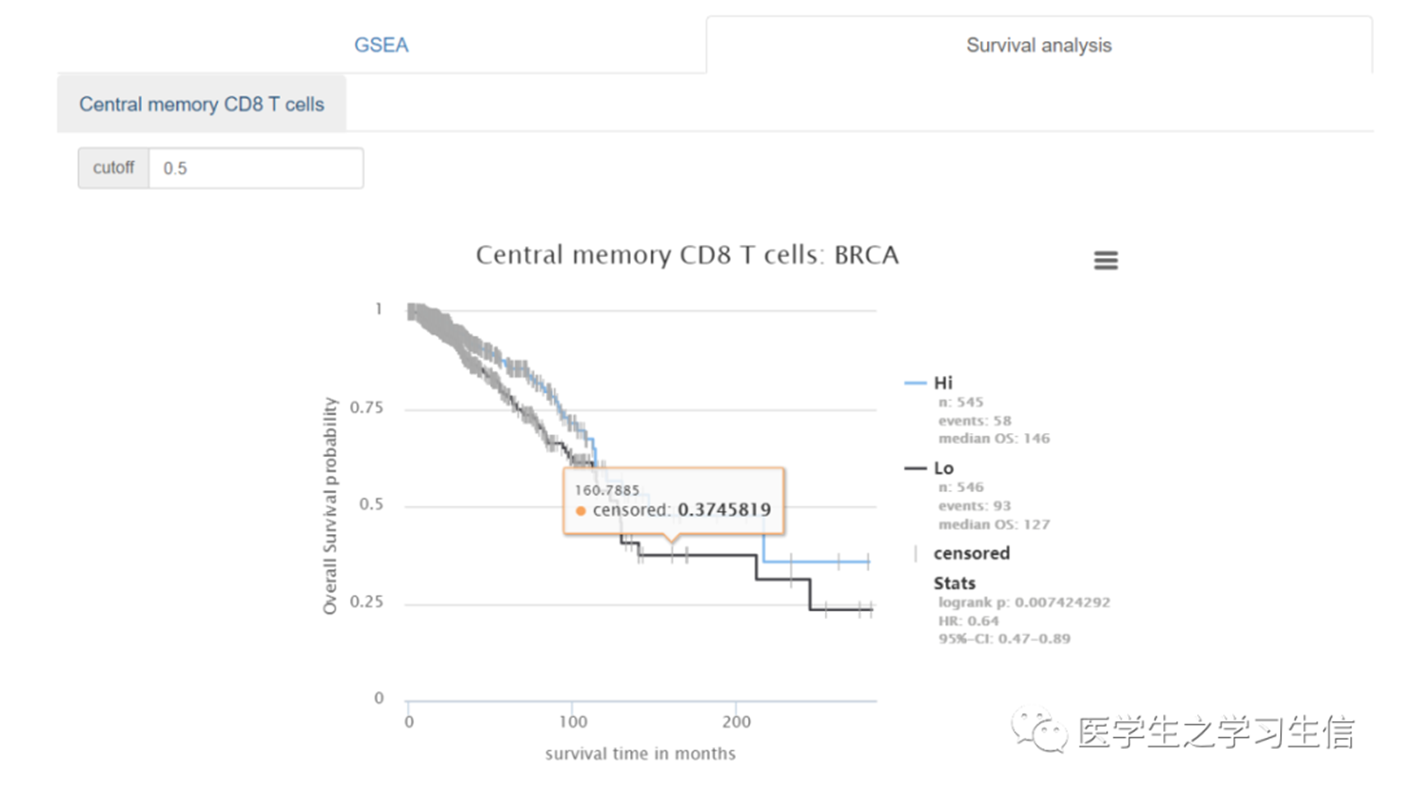
04 GSEA
这个模板提供我们常见的GSEA富集分析及生存曲线的绘制。
 与前面不同的是,这里要先选择细胞的种类↑
与前面不同的是,这里要先选择细胞的种类↑
05 Heterogeneity
TCIA对每个样本的癌症抗原和遗传特征进行了分析,包括肿瘤异质性和克隆性,以方便分析免疫特征和肿瘤的遗传特征。
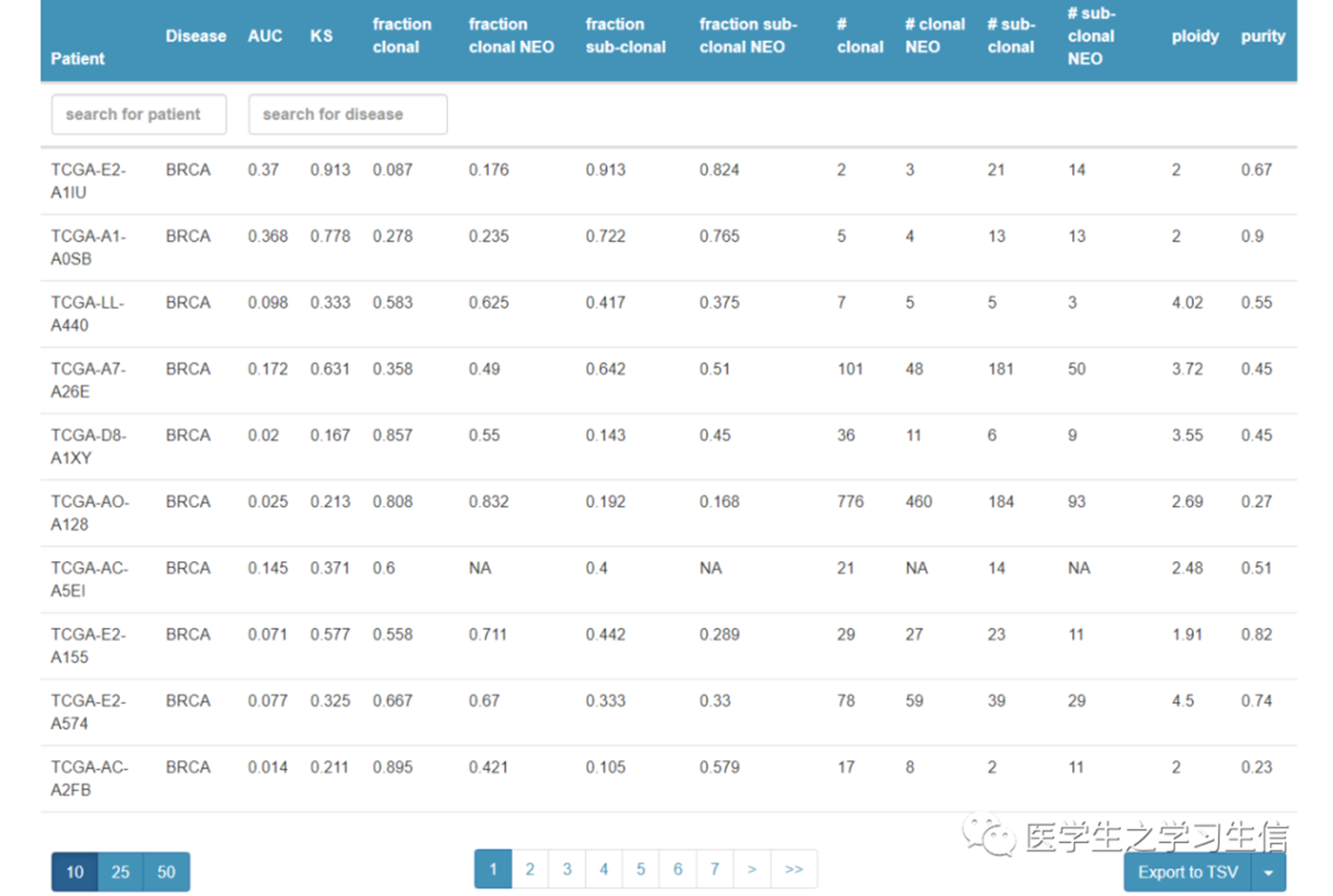
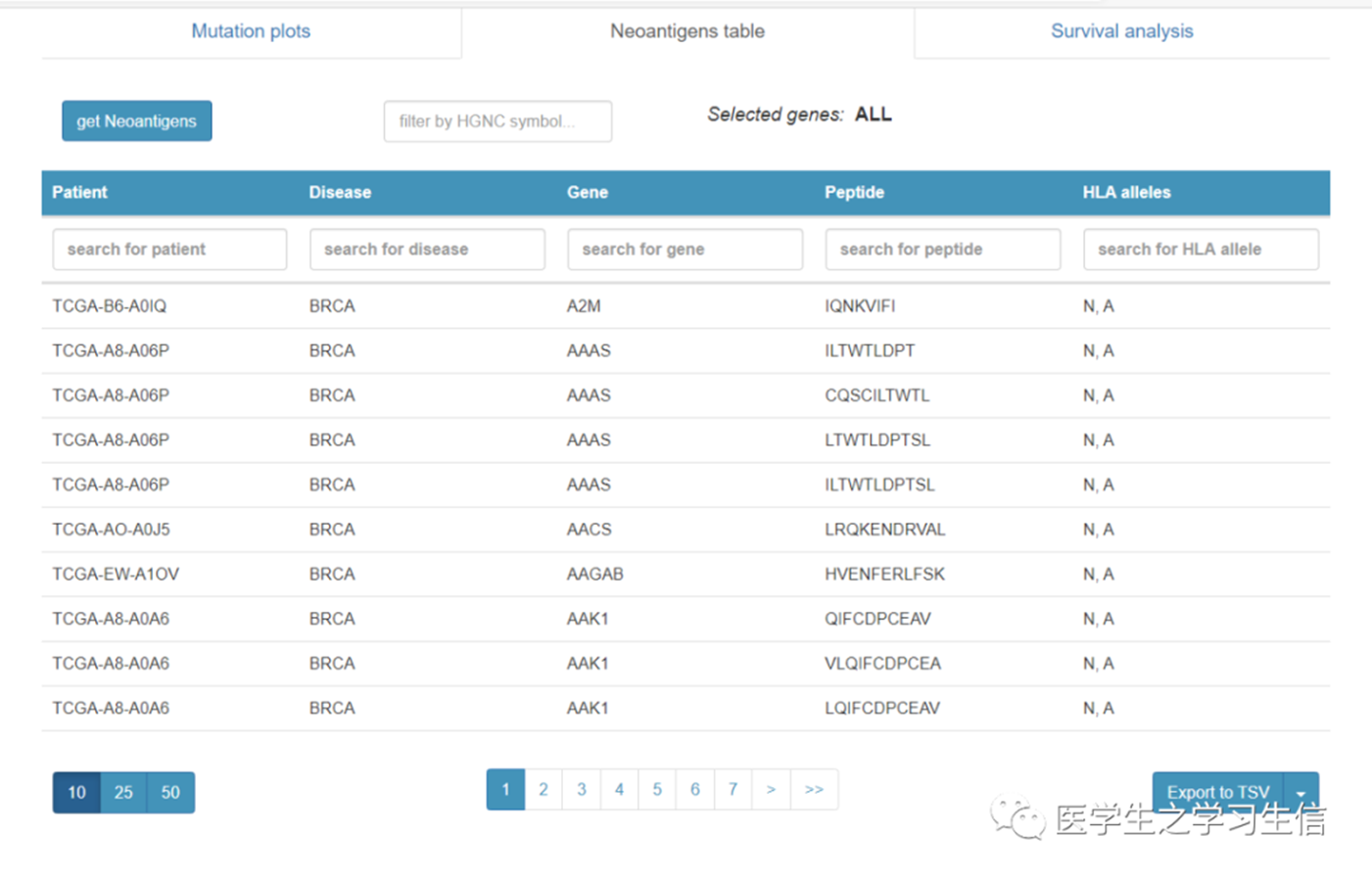
06 Neoantigens
这个模块提供疾病的突变数和突变压力展示,也提供展示预测到的新抗原结果。
(需要注意的是,新抗原预测因为没有办法用实验的方法证伪,所以仅仅用作分析还是可以的。)
07 Tools
这个模块提供一个计算功能,可计算每个样本的免疫表型分数,只要提供一个标准的表达矩阵即可,不过值得提醒的是该工具要求表达数据要求用TPM方法定量的。
 点击Home回到首页就能看见旁边有一个Tools的模块↑
点击Home回到首页就能看见旁边有一个Tools的模块↑

总的来说,TCIA数据库主要是对TCGA数据进行二次分析,得到的一些结果也可以辅助我们进行分析,它提供的IPS计算一定程度上也可以给NGS数据分析一些帮助。
本文转载自《医学生之学习生信》。如有侵权,请联系删除。