简介
dbNSFP,用于对人类基因组中所有潜在的非同义单核苷酸变异(nsSNV)进行功能注释和预测,前者提供与其潜在功能间接相关的突变的定性或描述性注释,例如突变是否为非同义SNV;后者通常基于统计模型提供突变的直接定量或是或否有害性预测。目前该数据库更新到了v4版本(每更新一个版本就发一篇文章),共包括了84,013,490个nsSNV和ssSNV。主要使用38种预测算法来对突变位点进行功能预测和9种保护打分算法:
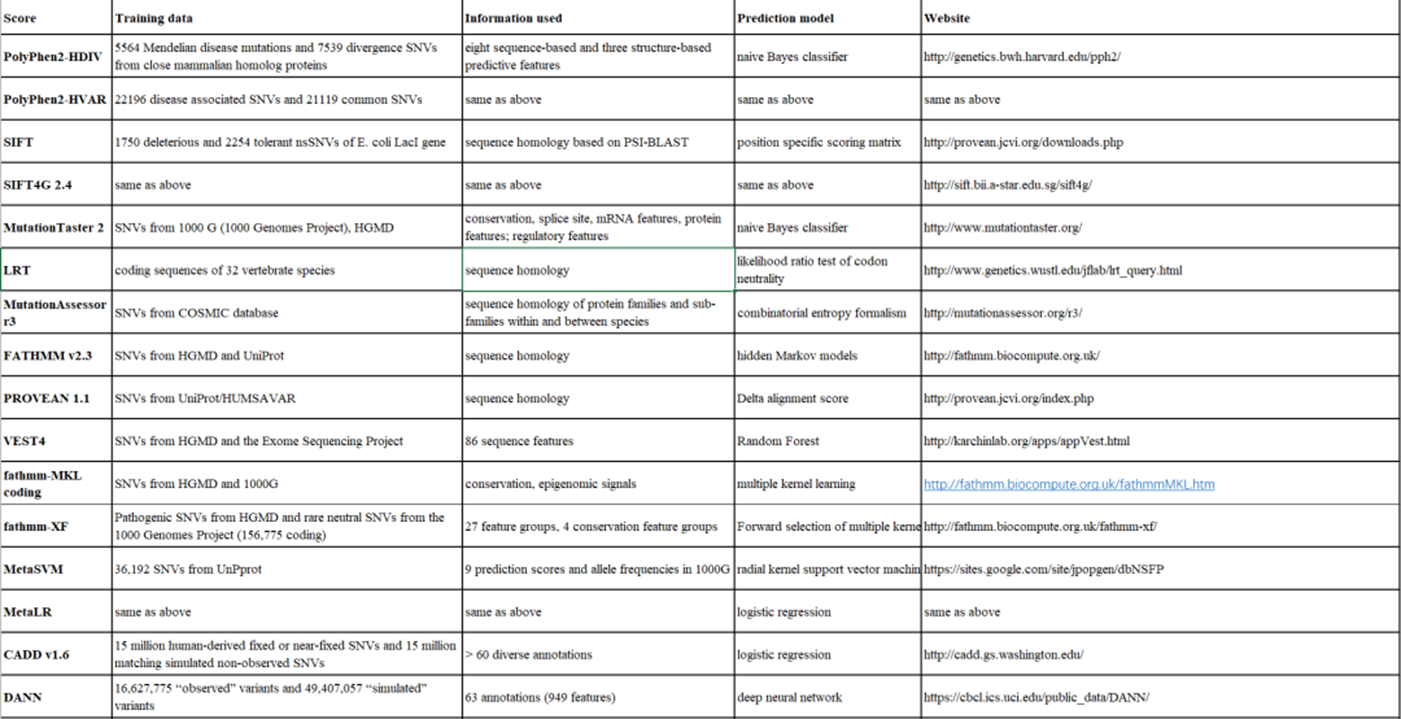
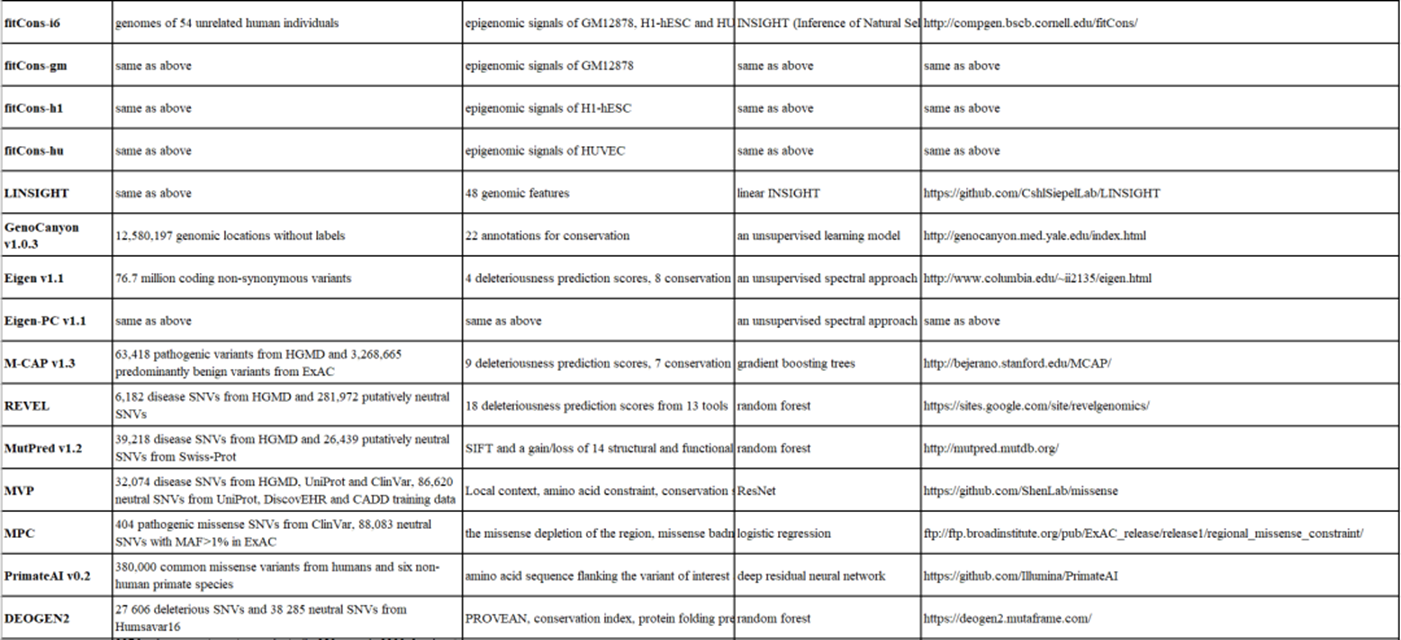
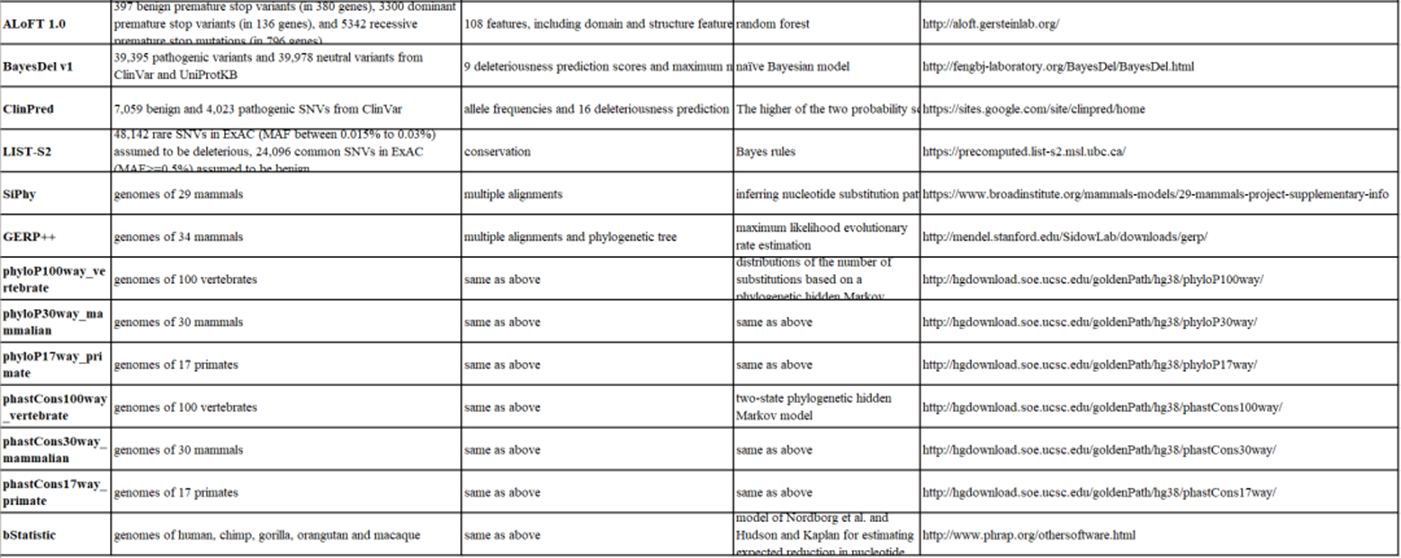
使用方法
该数据库提供了一个网页界面给用户访问:http://database.liulab.science/dbNSFP。不过区别于学术版和商业版,前者注释更为全面。
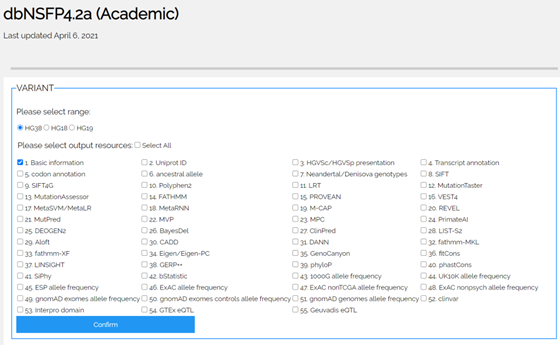
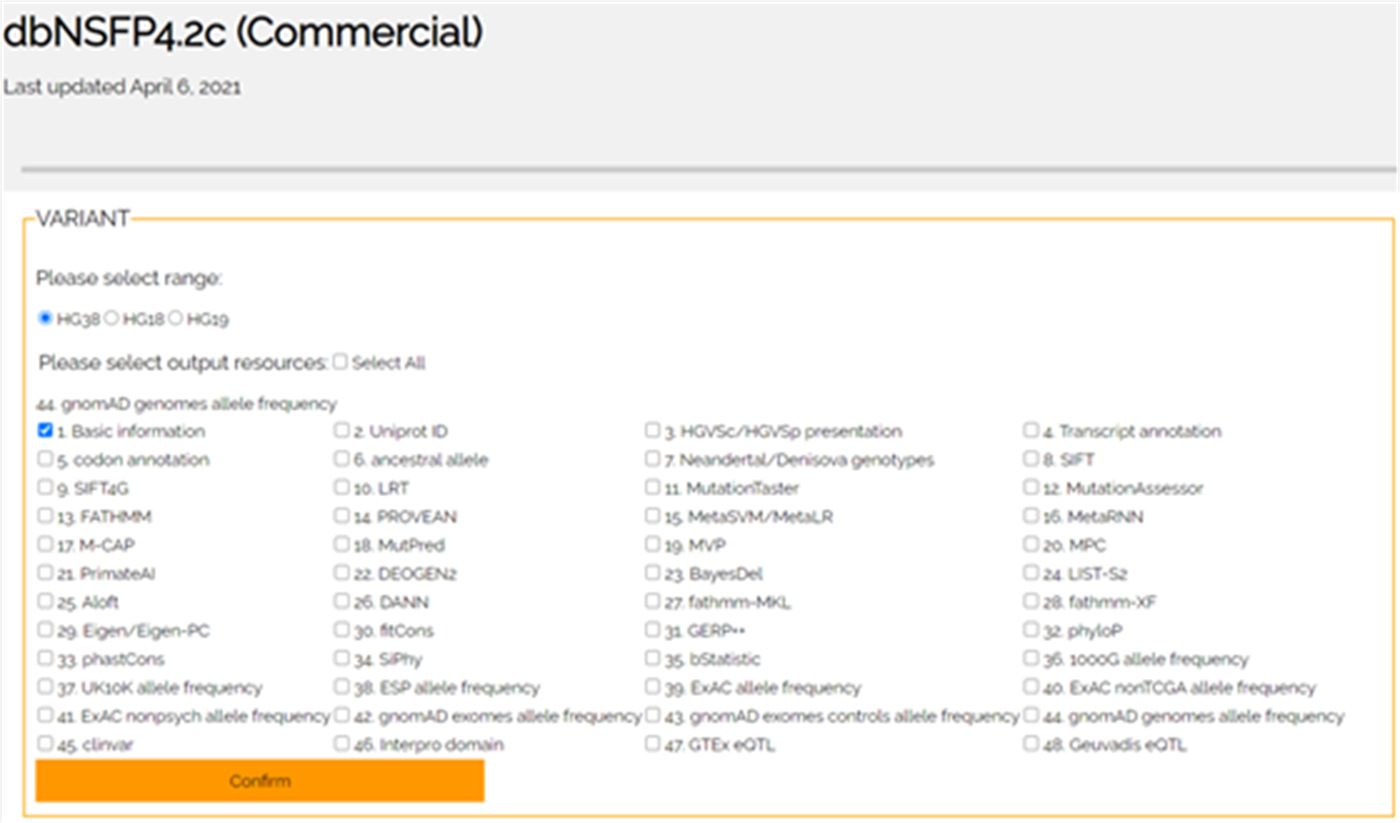
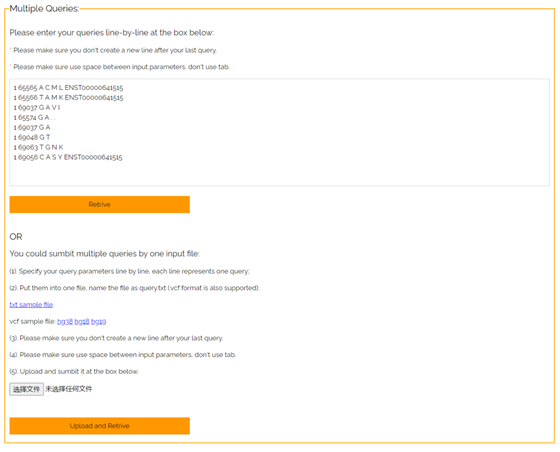
如果是小规模的数据,可以使用web服务进行突变的注释,只需要输入突变位点所在的基因组坐标(染色体、位置、参考和替代碱基),支持单个位点、多个位点或者上传vcf文件进行注释。

如果是大规模的数据,则需要下载数据库文件进行注释,在https://sites.google.com/site/jpopgen/dbNSFP提供了多种下载方式ftp、Google drive and Box,注释文件分学术版(v4.2a)和商业版(v4.2c),有30G左右,国内用户访问或下载较为困难。该链接还说明了不同更新版本的修改内容(即历史更新)。
也可以不下载该数据库,因为很多注释工具也会将该数据库的部分内容注释上,如variant tools, ANNOVAR, KGGSeq, VarSome, UCSC Genome Browser's Variant Annotation Integrator, Ensembl Variant Effect Predictor, SnpSift and HGMD。
相关文章:
1. Liu X, Jian X, and Boerwinkle E. 2011. dbNSFP: a lightweight database of human non-synonymous SNPs and their functional predictions. Human Mutation. 32:894-899.
2. Liu X, Jian X, and Boerwinkle E. 2013. dbNSFP v2.0: A Database of Human Non-synonymous SNVs and Their Functional Predictions and Annotations. Human Mutation. 34:E2393-E2402.
3. Liu X, Wu C, Li C, and Boerwinkle E. 2016. dbNSFP v3.0: A One-Stop Database of Functional Predictions and Annotations for Human Non-synonymous and Splice Site SNVs. Human Mutation. 37:235-241.
4. Liu X, Li C, Mou C, Dong Y, and Tu Y. 2020. dbNSFP v4: a comprehensive database of transcript-specific functional predictions and annotations for human nonsynonymous and splice-site SNVs. Genome Medicine. 12:103.
本文转载自《生信菜鸟团》。如有侵权,请联系删除。