水稻常用数据库介绍
近几年随着测序数据量呈井喷式增长,涌现出一大批优秀的数据库。而水稻和拟南芥分别作为植物研究中单双子叶的模式植物,其基因组信息得到了很大程度的挖掘。下面我结合自己对水稻基因组数据库两年多的使用经验,向大家介绍一下常用的水稻基因组数据库。如有不全之处还望大家多多指正。
1. RAP-db
我们知道水稻有籼粳之分,粳稻由于其本身抗性较好,遗传转化较为容易等优点,受到了科研人员的青睐,粳稻中的代表性品种当属日本晴。这个网站最初由日本人创立,并且每年都会有更新。你可以在这个网站使用GBrowse查看基因的分布、根据基因号或简称查找基因的功能和序列、使用blast搜索同源基因、使用ID Converter来转换基因号等等。最近他们更新了籼稻「Kasalath」的序列,对基因分化感兴趣的伙伴可以去挖掘一下。
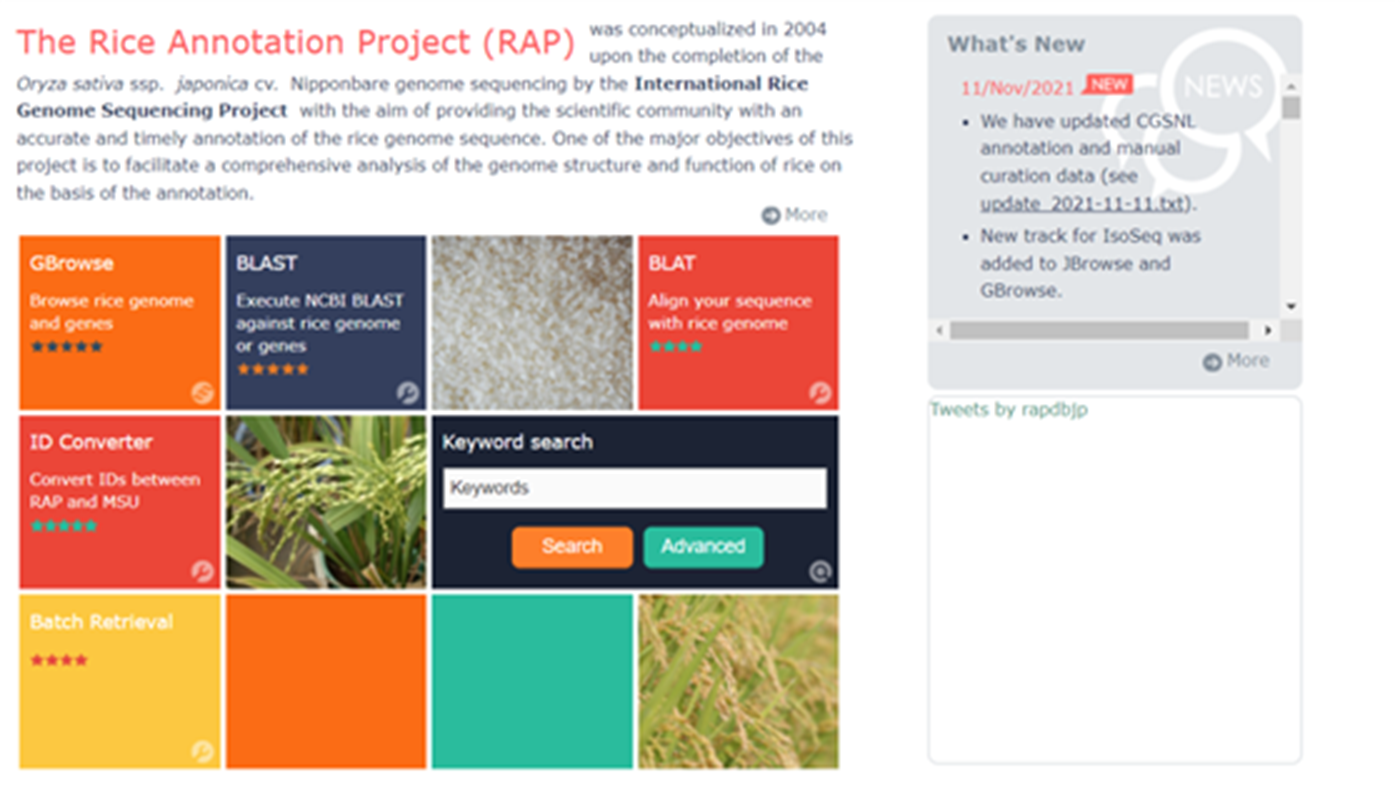 rap-db
rap-db
2.MSU-RGAP
RGAP是另一个受人们青睐的粳稻数据库,也是以日本晴作为参考基因组来注释的。最开始由密歇根州立大学牵头(最近他们似乎搬到了Georgia),通过gene ID可以和RAP-db进行基因名的转换,还可以进行go和kegg的搜索。(近几年的水稻基因组高分文章基本上都是采用的它的命名规则,我也不知道为何)
 msu
msu
3.籼稻注释网站RIGW
与粳稻相比,籼稻的种植面积更广,遗传多样性也更高。因此国内由华中农大牵头,对优良的籼稻品种珍山97(ZS97)和明恢63(MH63)构建了高质量的参考基因组,提供基因组学,转录组学,蛋白质–蛋白质相互作用(PPIs),代谢网络,代谢物和计算工具等,并且还可以做CRISPR靶点的选择。
4.整合数据库Gramene
水稻不仅籼粳分化严重,并且同一亚种间的分化也很严重,Gramene把人们研究较多的亚种各挑出一个代表来作为指示。Gramene是一个开源的,通过比较功能基因组学整合数据资源的网站。这里面罗列了不同水稻亚种的基因组信息,包括普通野生稻Oryza rufipogon,印度野生稻Oryza nivara,非洲栽培稻Oryza glaberrima等等。里面有一个功能我觉得对做信号传导的朋友有所帮助,称为「Plant Reactome」,它会把报道的基因的信号网络串联起来,形成信号网络,如生长发育信号、次生代谢途径、生物或非生物胁迫等。当然了,这和大牛写的综述是大同小异的,不同的是你可以在网站看到具体的数据,而不仅仅是基因上下游的关系。
5. 3K Rice Genome Project
由作科所牵头,对世界范围的3000余份水稻进行了重测序分析,构建了最大的水稻变异信息数据集,对水稻基因组学研究和育种应用提供了丰富的变异资源。
Data availablehttps://registry.opendata.aws/3kricegenome/
SNP-Seek:http://snp-seek.irri.org
交大版本:https://cgm.sjtu.edu.cn/3kricedb/index.php
6. Rice RC
Rice RC:http://ricerc.sicau.edu.cn/
由四川农业大学牵头,利用nanopore、转录组、Hi-C等技术构建了33个高质量的水稻基因组,并且挖掘出其中的结构变异和拷贝数变异,用来解释作物的驯化、产量等差异,对促进水稻育种和功能基因组学的研究具有重要的意义。
7.R498 & ZH11
Website:http://mbkbase.org/R498/
主要记录了籼稻R498和粳稻ZH11的基因组信息,但是从2018年开始就没有更新了。
本文转载自《生信媛》,如有侵权,请联系删除