Characteristics of the Gut Microbiome and Its Relationship With Peripheral CD4+ T Cell Subpopulations and Cytokines in Rheumatoid Arthritis(Front. Microbiol, 03 February 2022)
一、微生物的研究目标究竟是什么?
微生物研究尤其是医学疾病类微生物群落研究,费尽心思采集样本、测定微生物组成、含量到底要达到哪种目标?微生物研究核心目标在于帮助我们找到用于区分疾病/健康分组的一个或多个指示物种集合,用于分析在特定疾病中微生物群落的特征,并将这些指示物种用于预测样本的疾病状态、健康程度等,可指导后续实验验证方向、辅助指导临床治疗。
围绕风湿性疾病的发病机制和临床诊疗新技术开展研究。基于16s RNA测序数据,调查了类风湿关节炎(RA)中肠道菌群丰度和多样性、(CD) 4+T细胞亚群、细胞因子水平和疾病活性之间的关系。研究表明细菌谱系的失衡和肠道菌群代谢的改变导致宿主免疫图谱的改变,从而导致RA发病。
二、该如何实现样本分析?
使用到哪些分析点配合完成这些研究目标呢?可以大致概括为:1.基础分析;2.差异分析、指示物种筛选;3.功能描述;4.临床相关性分析。
1.基础分析
主要分析点:物种组成分析(堆叠图、维恩图等)、α/β多样性分析(PCoA降维图、各类多样性指数箱线图等)。
分析目标:微生物群落类文章在基础分析部分大同小异,都需要展示样本中有哪些微生物,整体趋势如何等基础信息,常用堆叠图等展示各分类水平下物种组成情况,箱线图等展示各分组多样性指数上的组成与差异趋势。
在基础内容展示部分往往需要给出PCoA等降维图形结果展示分组之间样本菌群组成差异。
分析结果:alpha多样性分析结果显示,肠道微生物群在RA患者相比健康人存在显著差异并减少(如图1所示)。基于weighted-UniFrac距离主坐标分析表明RA和HC组之间的肠道微生物群落结构不同(图2A所示)。
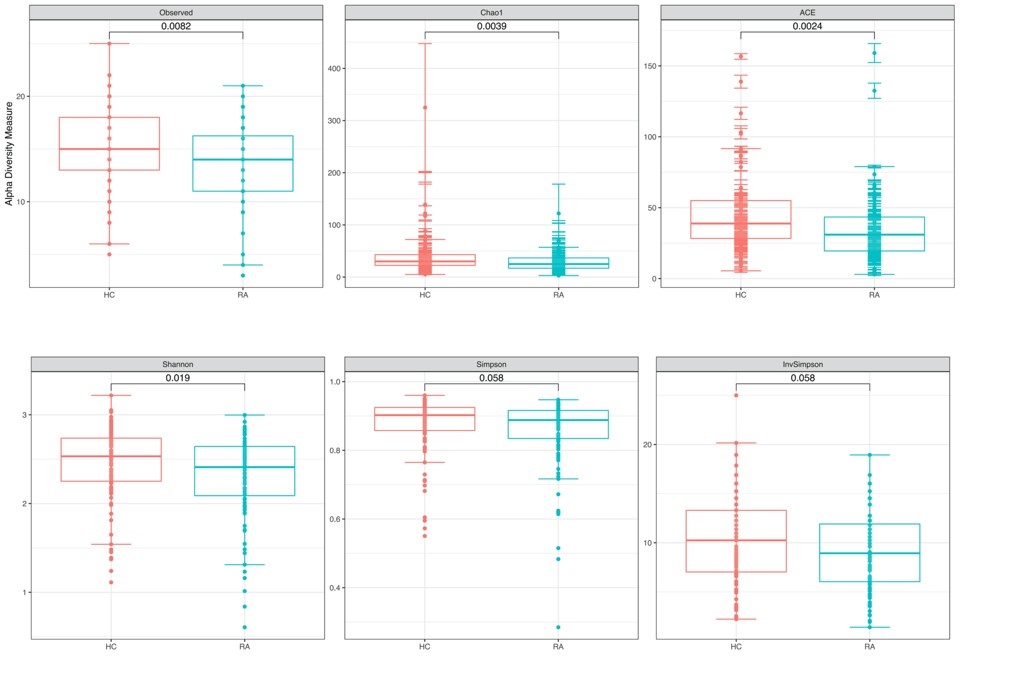
图1:Differences in αlpha diversity of the gut microbiome between RA patients and HCs.
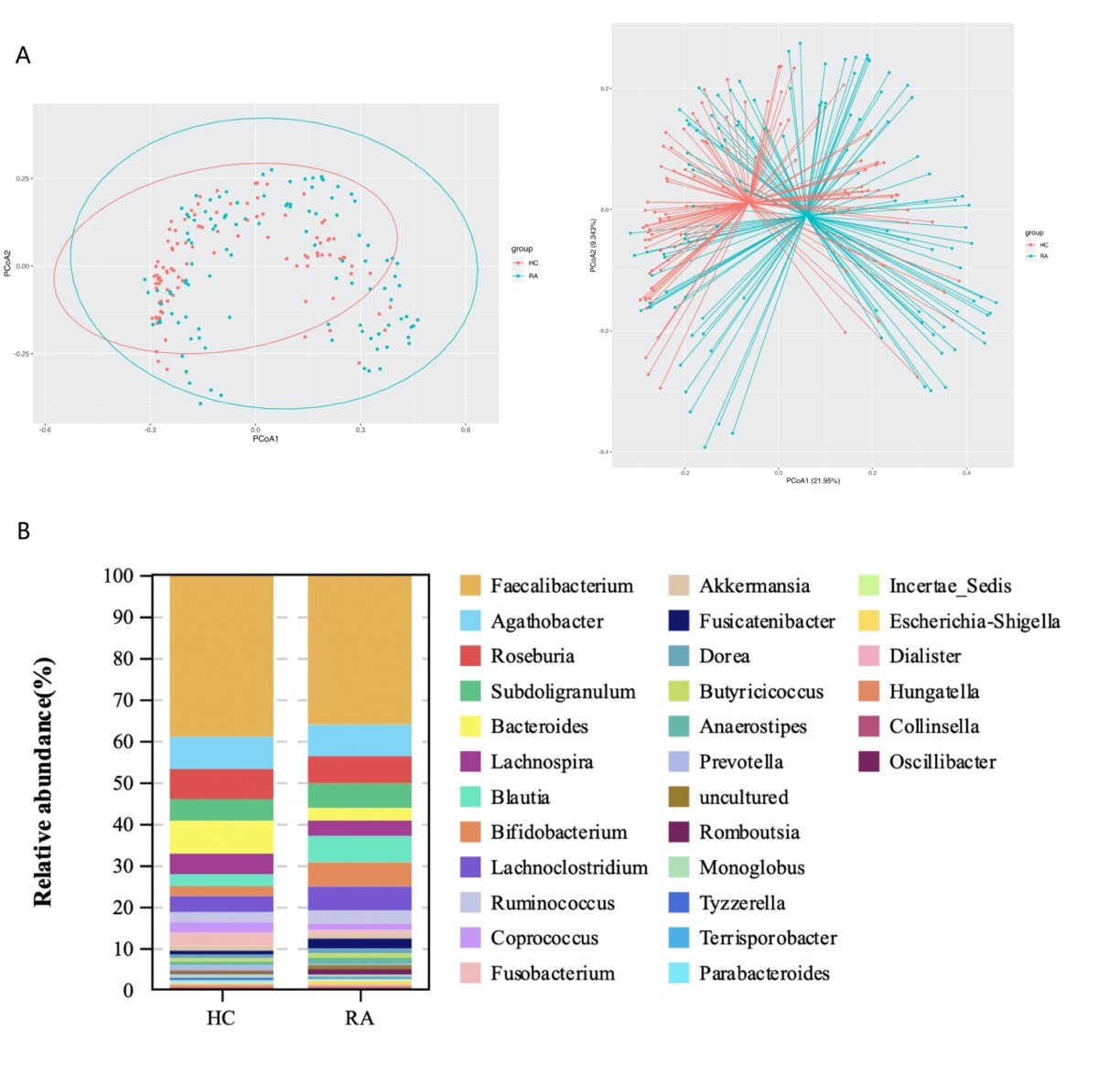
图2:(A)Beta-diversity of the gut microbiome in RA patients and HCs. Principal coordinate analysis plot generated from the weighted UniFrac analyse.(B)Microbiota community composition of RA patients at the phylum level.
之所以需要对分组进行展示与检验,原因在于病例对照研究以及各类队列研究中往往将微生物定义为一种暴露因素,疾病/健康状态作为结局。而在实际分组时并不能准确界定哪些微生物是暴露因素而是将疾病/健康状态作为分组依据进行样本划分,因此在结果中需要用PCoA等图形进行初步分型,描述分组方案可信。
2.差异分析、指示物种筛选
主要分析点:lefse分析
分析目标:第二阶段就需要针对物种进行筛选,找到组间具有统计学差异的物种,如常用的各类检验(T检验、方差分析等)、广受欢迎的lefse分析等都是可行方案(如图3的A、B所示)。
分析结果:通过LEfSe分析共观察到28个差异丰富的类群。
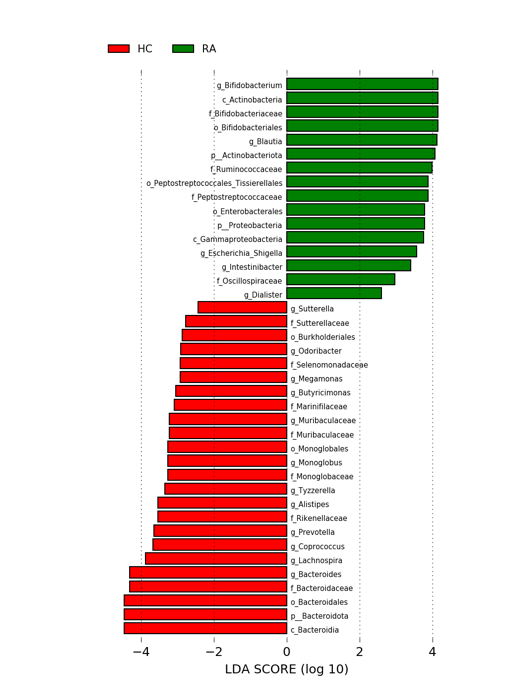
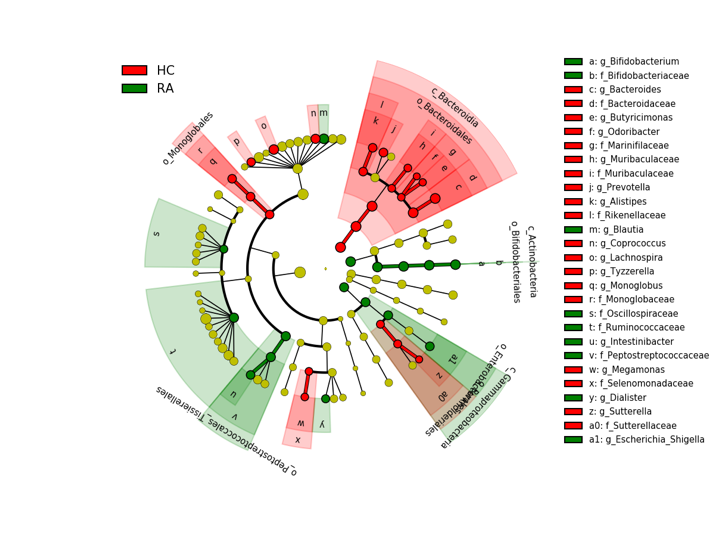
图3:Patients with RA characterized by expansion of rare microbial lineages. (A, B) LEfSe analysis was performed to identify differentially abundant taxa, which are highlighted on the phylogenetic tree in cladogram format (A) and for which the linear discriminant analysis scores are shown (B). Red and green colors indicate an increase or decrease in taxa, respectively, in RA patients compared to HCs.
3.功能分析
主要分析点:KEGG等数据库群落功能丰度输出。
分析目标:完成了指示物种筛选后物种的分析工作基本完成,紧接着需要对功能表型进行描述,从微生物群落角度出发,借助各类功能数据库进行功能丰度输出,挑选代表性通路描述组间功能差异是一条不错的思路(如图4所示)。
分析结果:RA患者与HC之间存在显著差异的类群的功能.
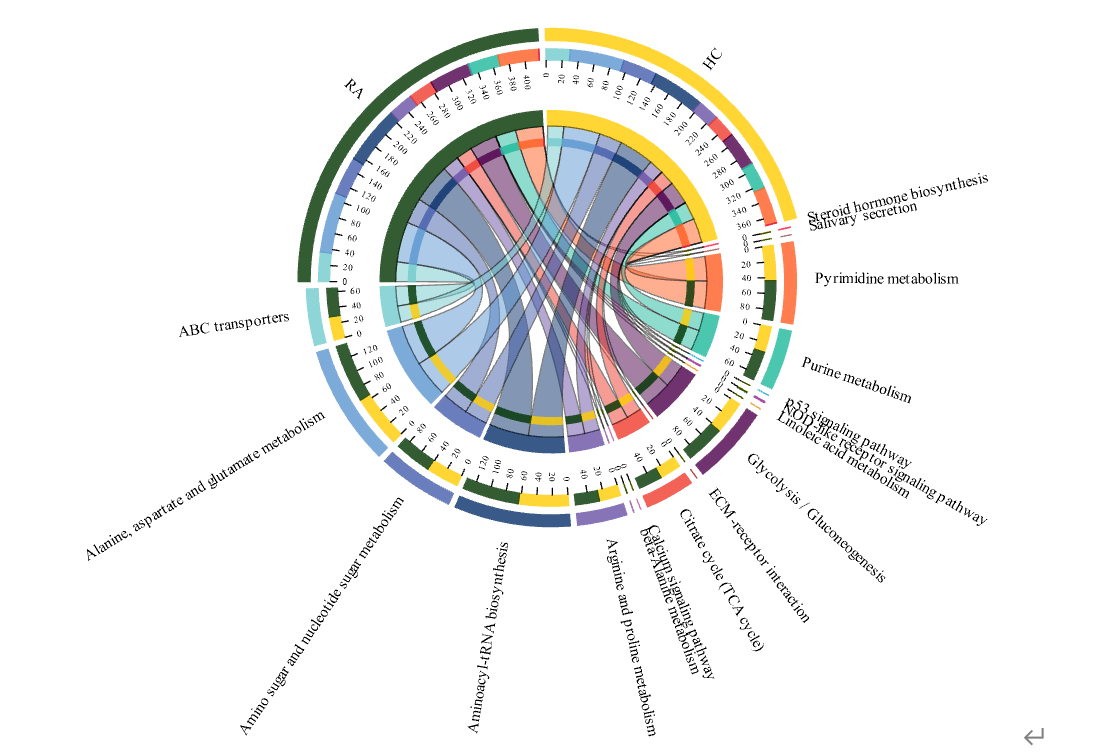
图4:KEGG pathways significantly enriched in RA patients compared to HCs.
4.临床相关性分析
主要分析点:肠道菌群与CD4+T细胞亚群及细胞因子水平的关系及疾病活性与肠道菌群、淋巴细胞亚群和细胞因子的关系。
分析目标:将这些指示物种用于预测样本的疾病状态、健康程度等,可指导后续实验验证方向、辅助指导临床治疗。
分析结果:与HC相比,RA患者的肠道微生物群与CD4+ T细胞计数、细胞因子水平和疾病活动性之间存在相关性,这证明了肠道微生物群在RA发病机制中发挥着重要作用。
三、结论
我们发现RA中肠道菌群组成发生了改变,这主要与代谢信号的变化有关,这可能导致CD4+T细胞亚群和细胞因子失衡,并影响疾病活动。这些发现为RA的可能致病机制提供了新的见解,并表明该疾病可能是通过调节肠道微生物群影响CD4+T细胞亚群和细胞因子来调节的。
四、小结
微生物高分文章中的指示物种挖掘,临床相关性研究有一套相对成熟的分析逻辑,体现在基础分析、指示物种探索、功能分析以及临床相关性分析。指示物种可以是一种或多种微生物的集合,需要用到lefse软件,除此之外,也可以使用随机森林、ROC曲线等传统分类器手段(也有些文章用到各类机器学习分类器方案)。
在确保筛选出的指示物种效果足够优秀的前提下,开展功能分析,对指示物种结论进一步深化与验证,之后结合实验类型针对性开展验证实验,即可完成一篇逻辑严密的围绕微生物对样本分析的群落文章。